Life Sciences Linked Data
Máster Bioinformática UM
Mikel Egaña Aranguren
http://mikeleganaaranguren.com / mikel.egana.aranguren@upm.es
Biological Informatics Group (http://wilkinsonlab.info), CBGP, UPM, Madrid
Life Sciences Linked Data
https://mikeleganaaranguren.wordpress.com/teaching/
http://biordf.org:8080/UM_LSLD/Clases/UM_Bioinformatics_LD.html

¿Qué es Linked Data?
Video divulgativo
¿Qué es Linked Data (LD)?
Un método para ofrecer datos directamente en la web
Una propuesta del W3C: http://www.w3.org/standards/semanticweb/data
Un primer paso hacia la Web Semántica
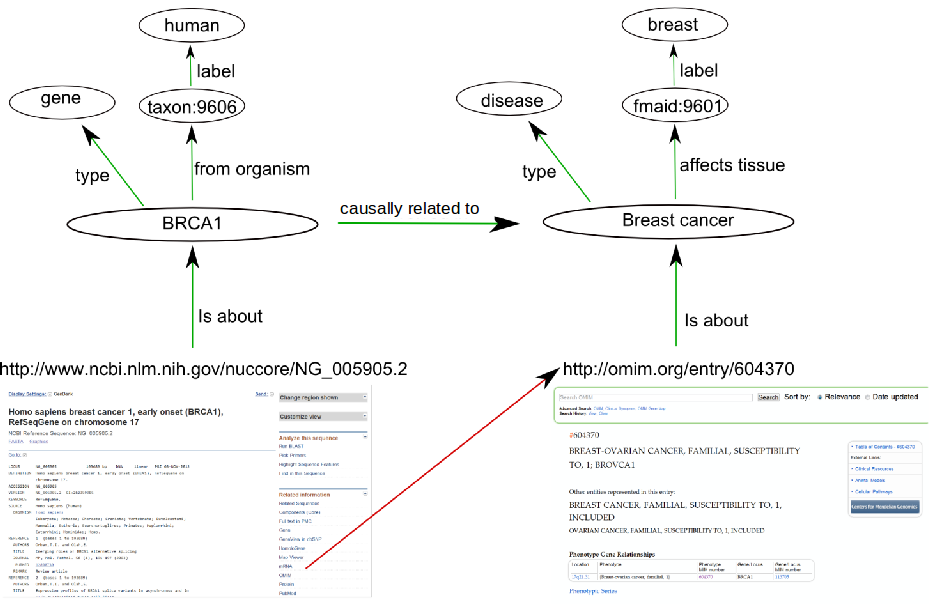
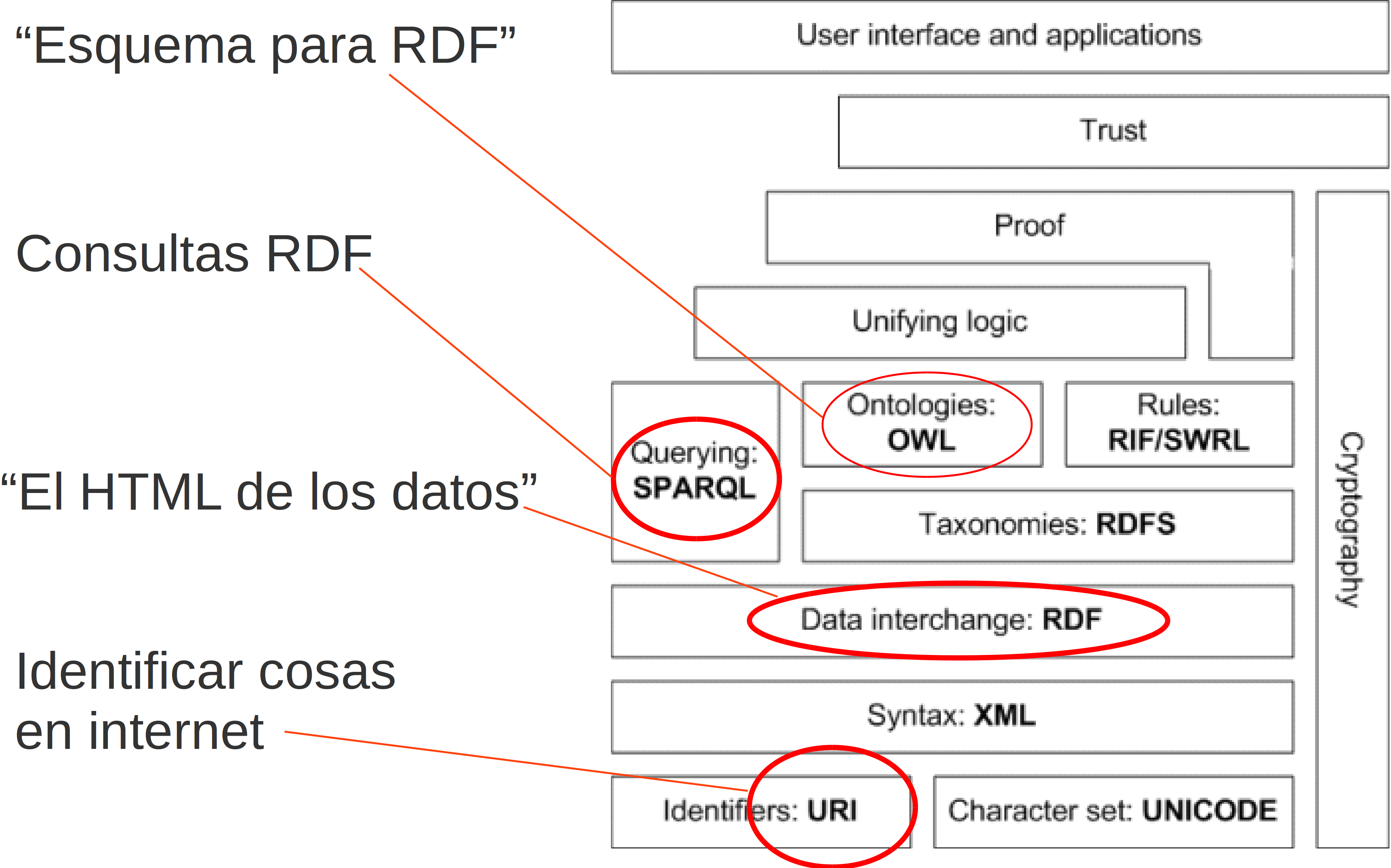
Pila tecnologías Web Semántica
Un primer paso hacia la Web Semántica
LD utiliza la tecnología ya existente (URI, HTTP, ...) para ofrecer una primera versión de la Web Semántica
Pila tecnologías Web Semántica y LD
Principios LD
- Usar URIs para identificar entidades
- Usar URIs HTTP para que se pueda acceder a esas entidades
- Cuando un usuario o agente accede a una URI, proveer información útil mediante estándares (RDF, SPARQL)
- Incluir enlaces a otras URIs para que se puedan descubrir más entidades
http://www.w3.org/DesignIssues/LinkedData.html
Con LD publicamos datos de manera ...
... semántica
... enlazada
Semántica
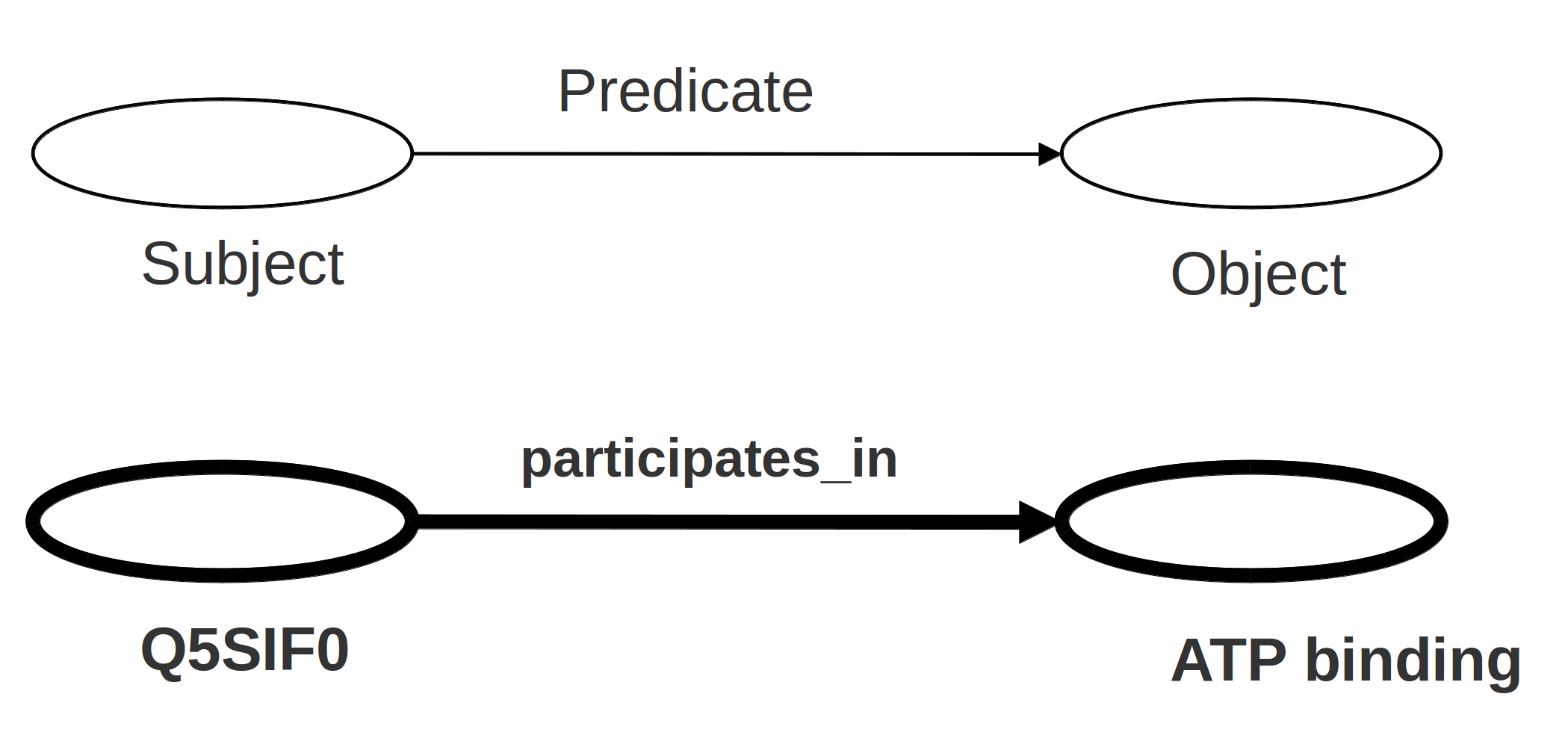
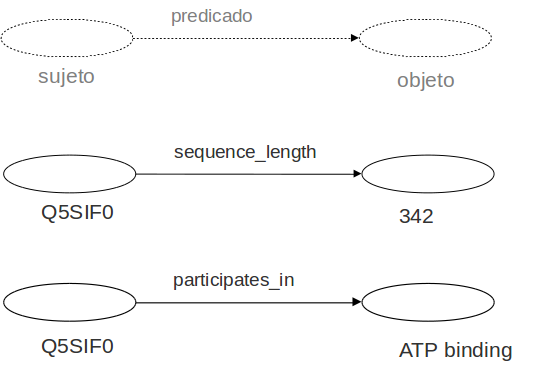
RDF ofrece el triple, un modelo de datos explícito y homogéneo: una "frase" estándar que los ordenadores pueden "entender"
Enlaces
En el triple, cada entidad (sujeto, predicado, objeto) tiene una URI que lo identifica
Los datos son enlazados a otros datos a través de la web, con enlaces explícitos
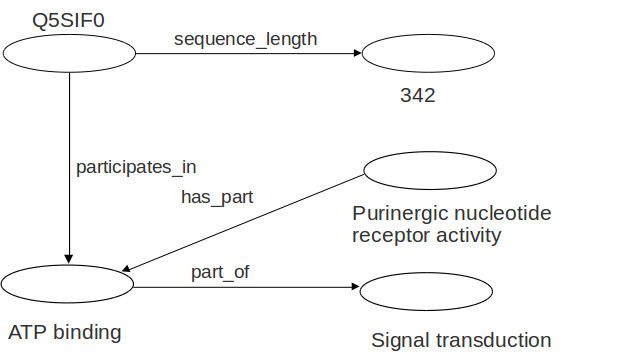
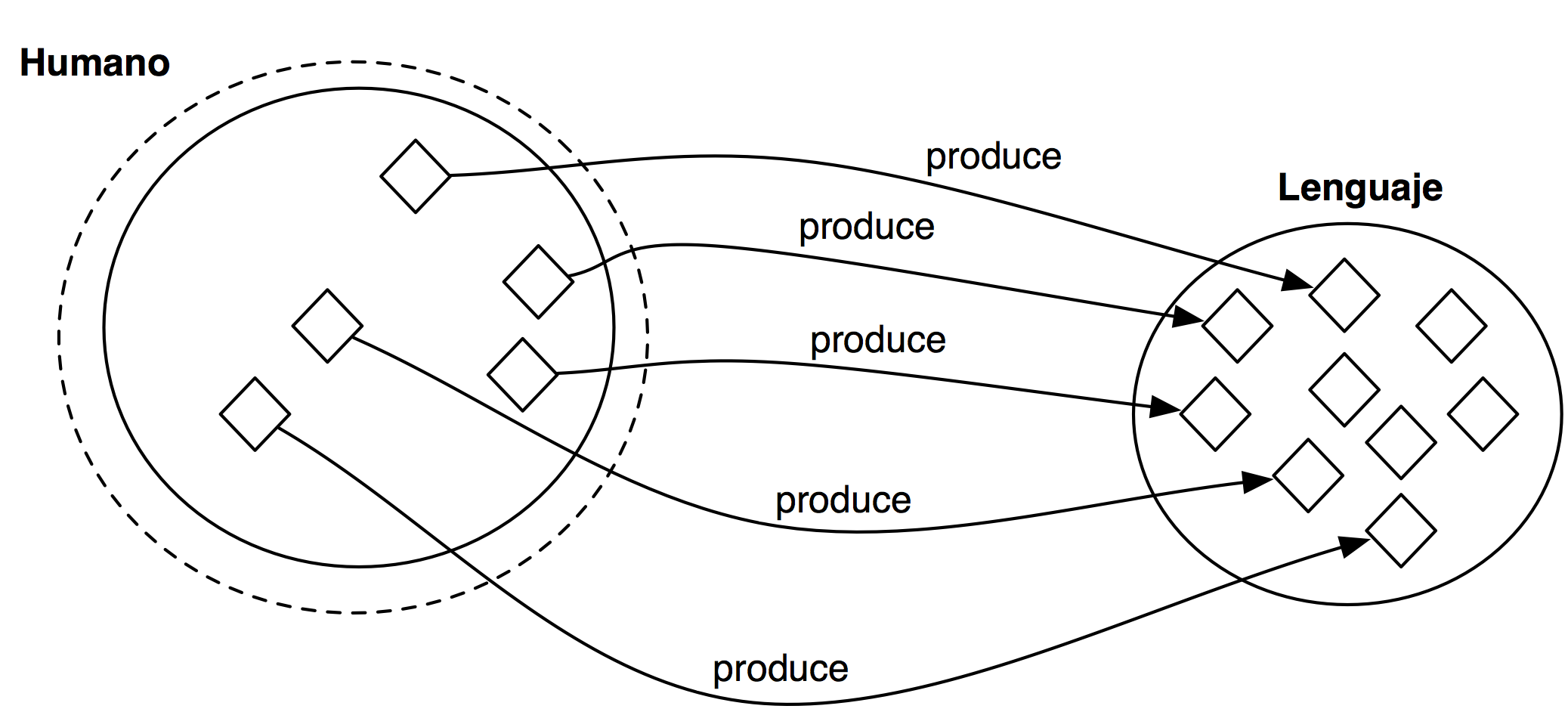
Red global de datos enlazados
Red global de datos enlazados
Internet de datos, en vez de documentos: "Base de Datos universal":
- Es más fácil construir aplicaciones que exploten los datos, incluyendo razonamiento automático
- Encontramos justo lo que buscamos: consultas directas (SPARQL) en vez de procesar texto
Red global de datos enlazados
Navegamos directamente por las datos (RDF), en vez de navegar a través de documentos que representan esos datos en lenguaje natural (HTML)
Enlazar datos nuevos es tan fácil como enlazar páginas web: crecimiento orgánico de la red
Linked Open Data (LOD) cloud
http://richard.cyganiak.de/2007/10/lod/
Datasets de interés
- Bio2RDF
- OGOLOD (¡UM!)
- LinkedLifeData
- HyQue*
- ArrayExpress and Gene expression atlas*
- UniProt
- LOD cloud ...
RDF
RDF (Resource Description Framework)
RDF es un estándar oficial del W3C para representar información en la web
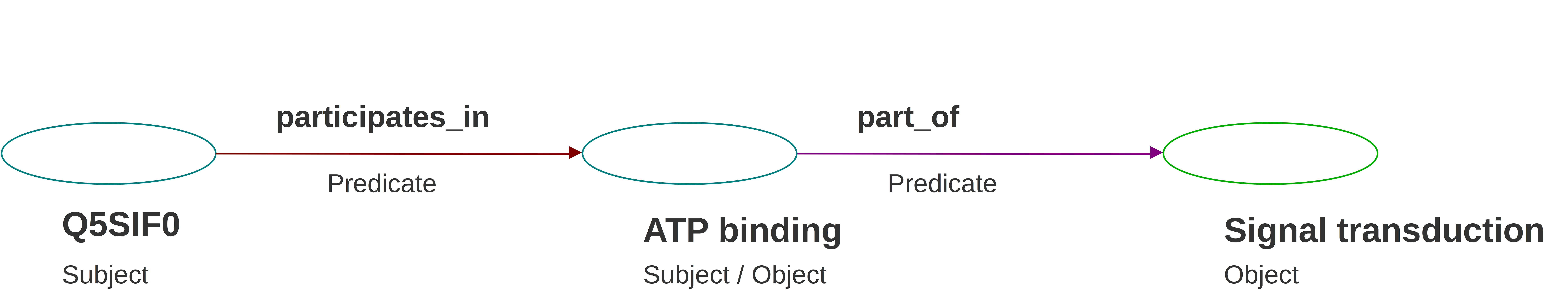
Triple RDF
Grafo RDF
Un grafo RDF es un conjunto de triples
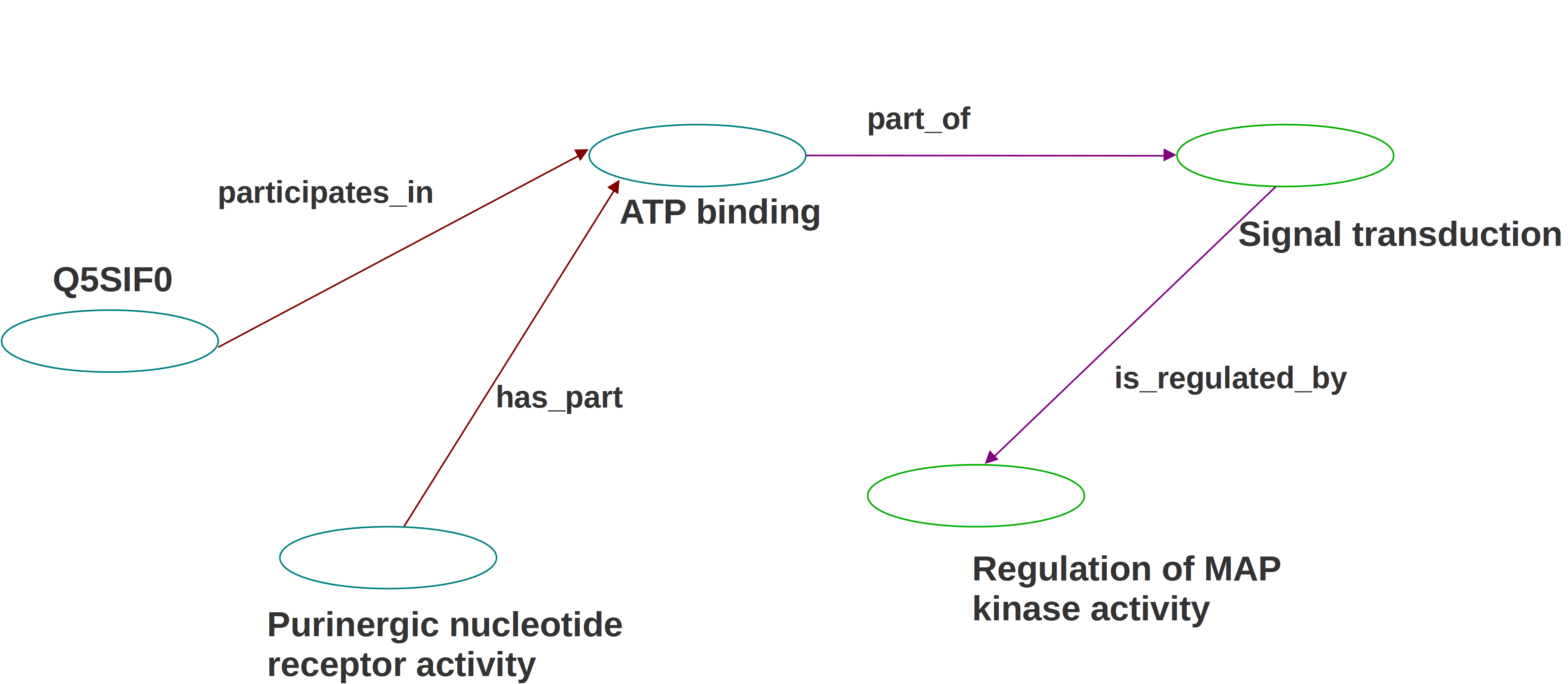
Grafo RDF
Algunos objetos pueden ser valores literales (Cadenas de caracteres)
Sujetos y predicados sólo pueden ser recursos
Los valores literales pueden tener tipo (XML Schema datatypes)
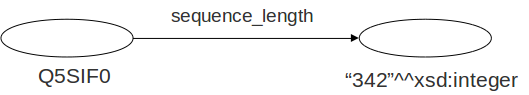
Elementos RDF
rdf:type: agrupar recursos en clases
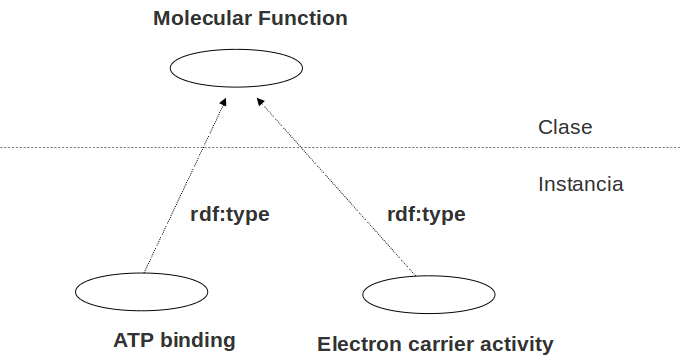
Elementos RDF
URIs en RDF
Cada recurso (Sujeto, predicado, objeto) tiene una URI
URI: Uniform Resource Identifier (RFC3986)
Reference URI: URI + fragment (http://foo/bar#frag)
(En Linked Data, las URIs se pueden resolver, es decir de-referenciar)
Espacios de nombres XML
RDF usa espacios de nombres XML mediante "qualified names"
Vocabulario: URIs bajo un espacio de nombre
- rdfs="http://www.w3.org/2000/01/rdf-schema#"
- obo="http://purl.org/obo/owl/GO#"
- owl="http://www.w3.org/2002/07/owl#"
- ...
Serializar RDF
RDF es un modelo para representar datos
Ese modelo abstracto se puede representar con diferentes sintaxis: "Serializar" (escribir) en un archivo
Una de esas sintaxis es RDF/XML
No confundir el modelo con la sintaxis: ¡RDF es mucho más que un archivo XML!
Serializar RDF
- RDF/XML (http://www.w3.org/TR/rdf-syntax-grammar/)
- RDFa (http://www.w3.org/TR/rdfa-core/)
- Turtle (http://www.w3.org/TR/turtle/)
- N3 (http://www.w3.org/DesignIssues/Notation3.html)
- ...
Serializar RDF: RDF/XML

Vocabulario (ontología) / triples
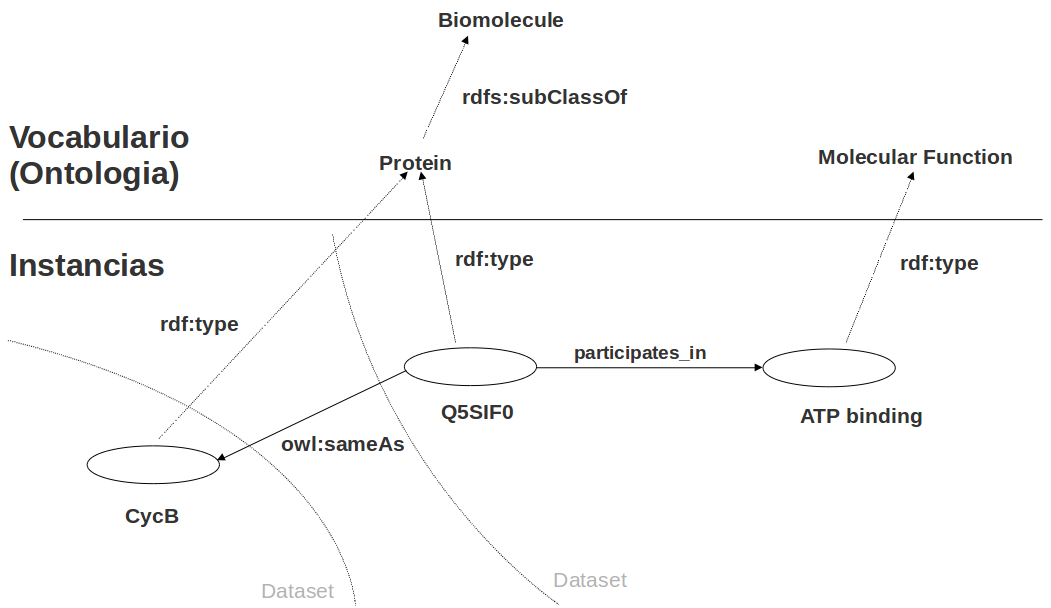
Más información
RDFS (RDF Schema)
RDFS
RDFS es un estándar oficial del W3C para describir clases de recursos RDF y sus propiedades: definir vocabularios para RDF
RDFS
OWL (Web Ontology Language)
OWL
OWL es un estándar oficial del W3C para crear ontologías en la web con un semántica precisa y formal
OWL
OWL se basa en Lógica Descriptiva (DL)
Representación computacional de un dominio de conocimiento:
- Razonamiento automático: inferir conocimiento "nuevo" (*), consultas, consistencia, clasificar entidades contra la ontología, ...
- Integrar conocimiento disperso
Sintaxis OWL
Para ordenadores: RDF/XML, OWL/XML, ...

Para humanos: Manchester OWL Syntax, functional, ...

Semántica OWL
Una ontología OWL esta compuesta de:
- Entidades: las entidades del dominio de conocimiento, identificadas con URIs, introducidas por el desarrollador ("proteina", "participa_en", ...)
- Axiomas: relacionan las entidades mediante el vocabulario lógico que ofrece OWL
Una ontología puede importar otra (owl:import) y hacer referencia a sus entidades mediante axiomas
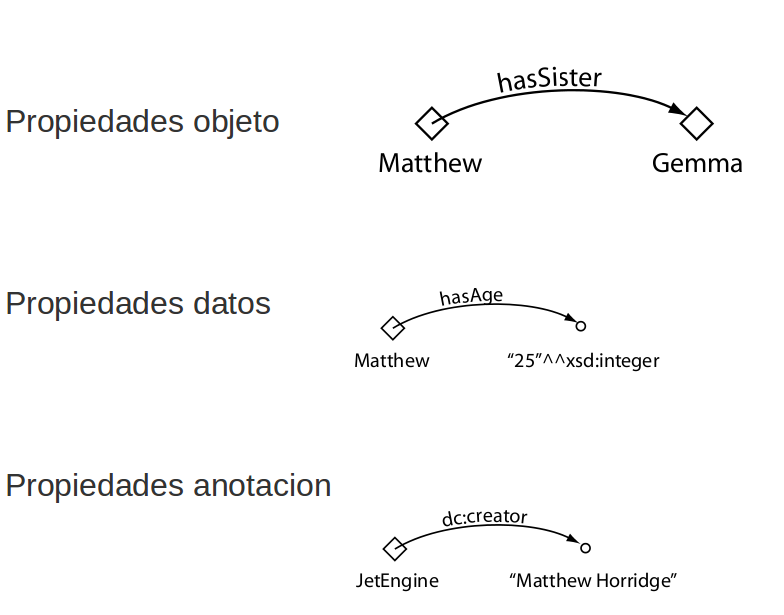
Entidades OWL
- Individuos
- Clases
- Propiedades
- Objeto
- Anotación
- Datos
Individuos
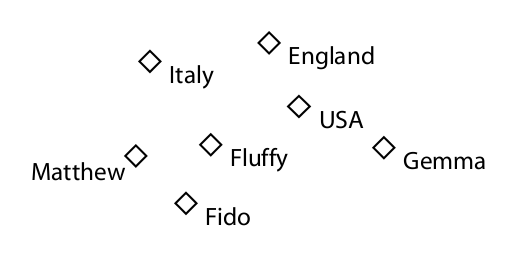
Propiedades
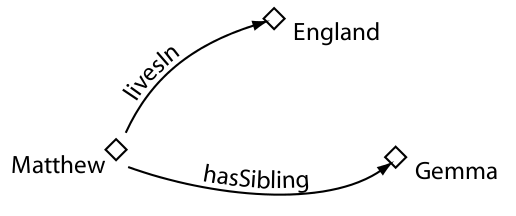
Clases
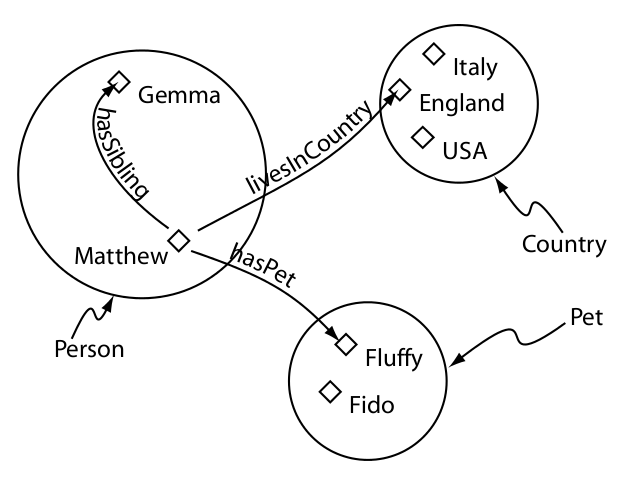
Semántica OWL
Una ontología OWL formada por individuos y clases es una "Base de Conocimiento" (KB) formada por:
- TBox (Terminological Box): clases (~ "esquema")
- Abox (Assertional Box): individuos (~ "datos")
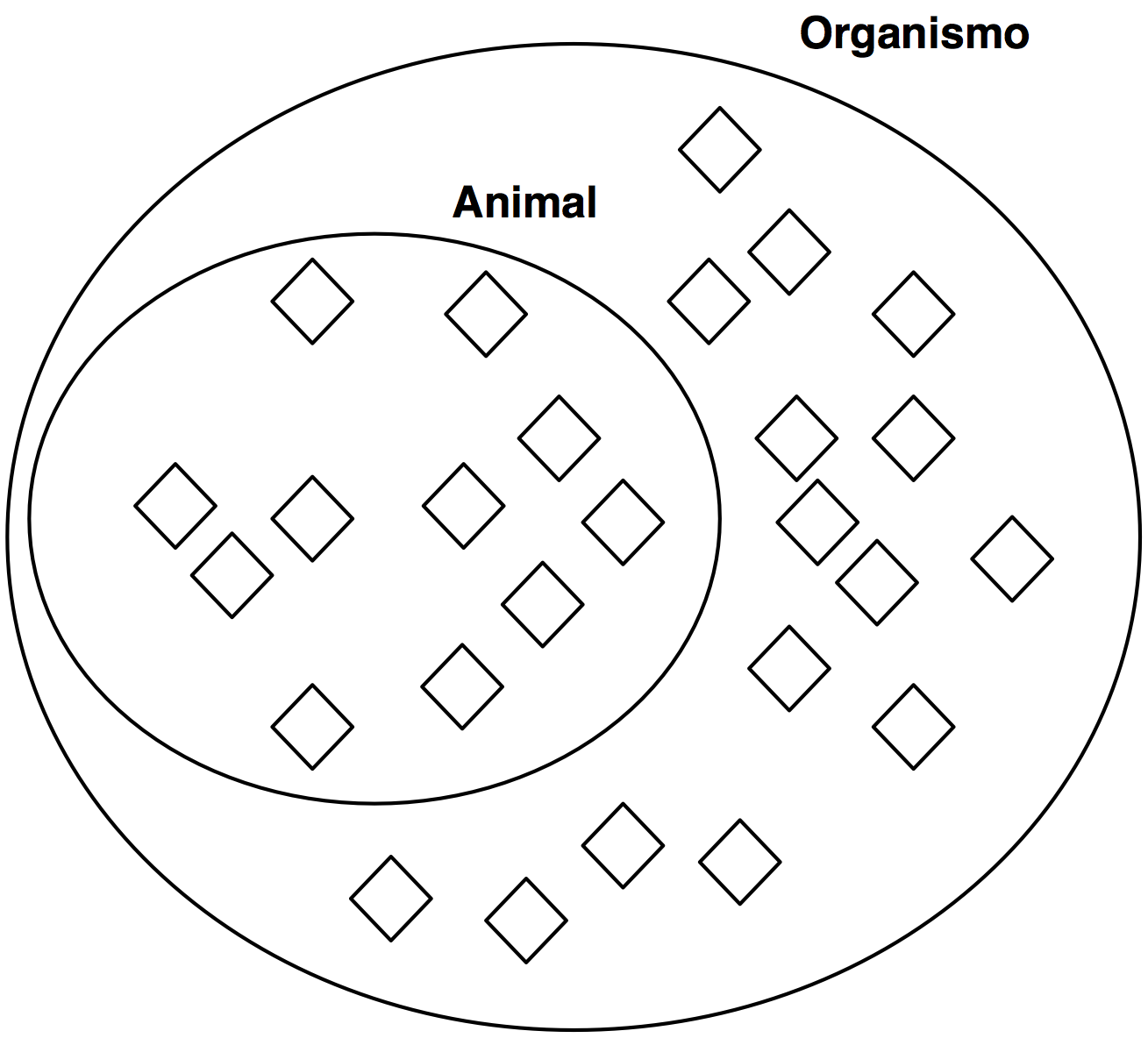
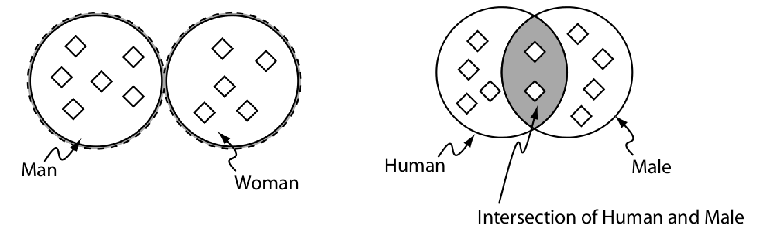
Clases
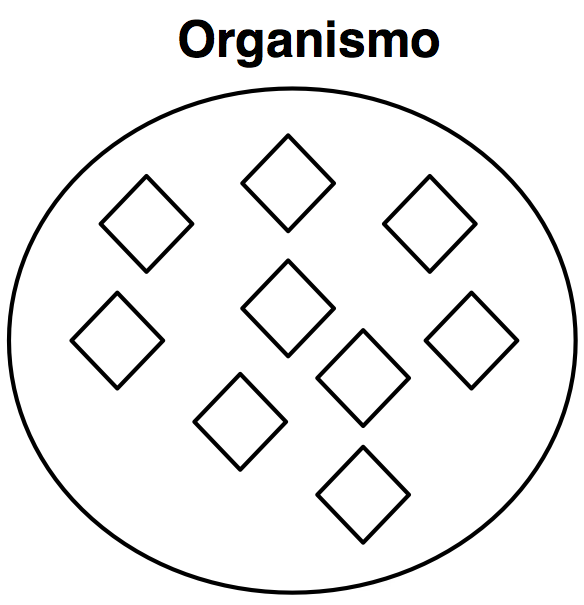
Clase subclase
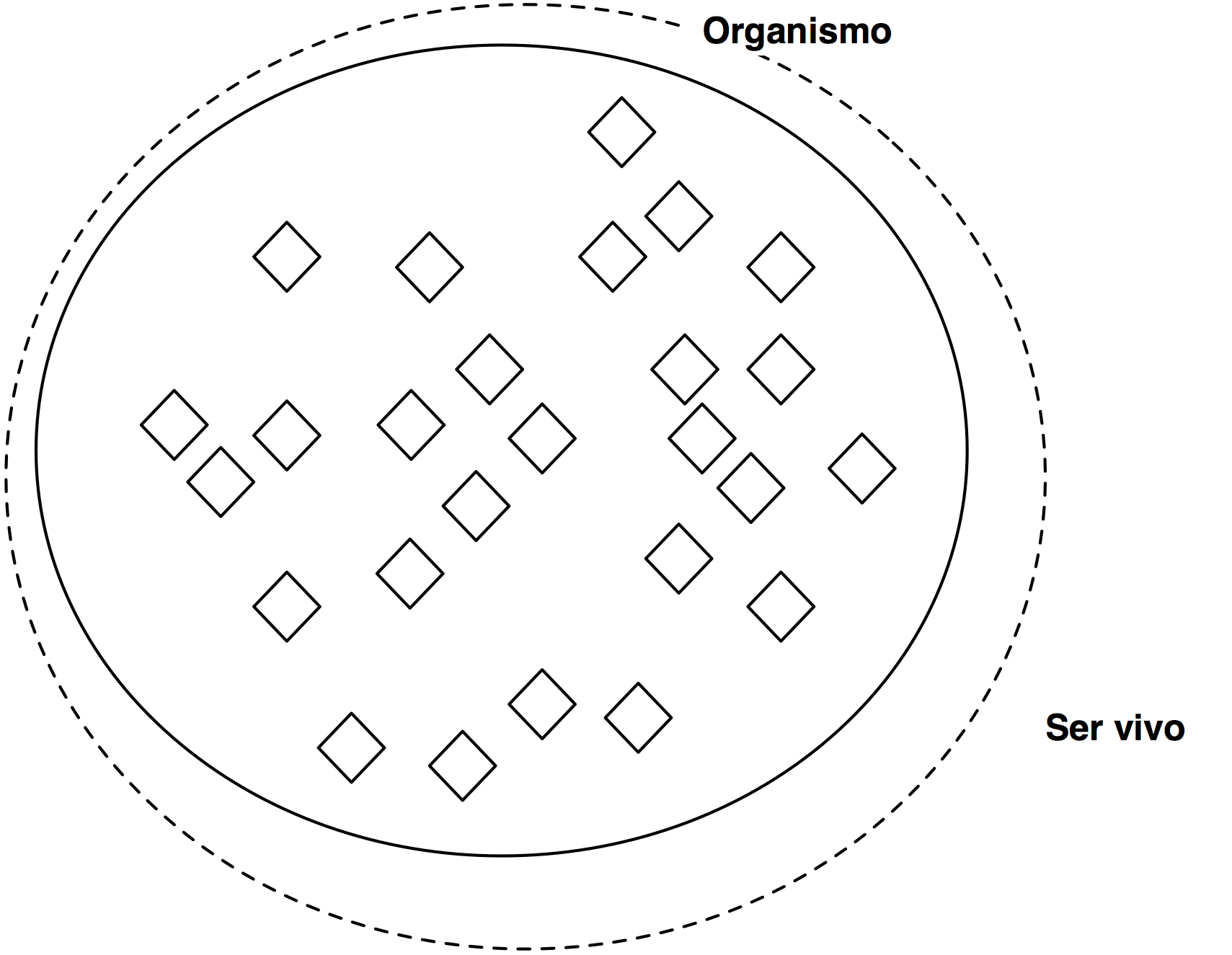
Clases equivalentes
Jerarquía de clases (Taxonomía)
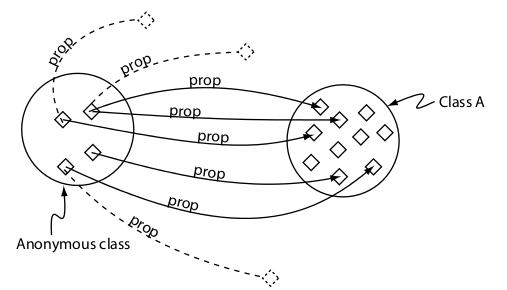
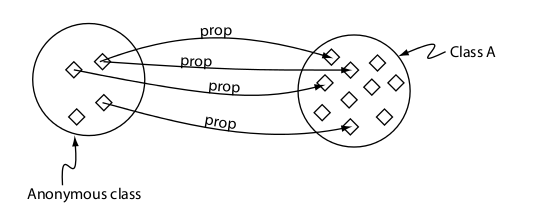
Condiciones necesarias
Condiciones necesarias y suficientes
Restricción existencial (some)
Restricción universal (only)
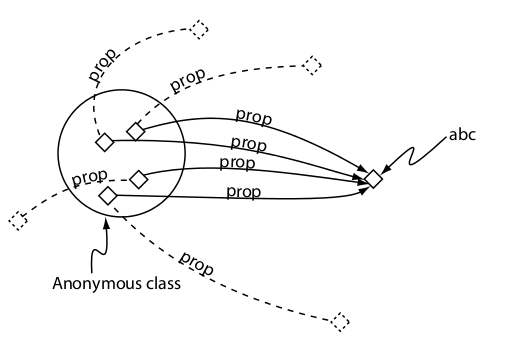
Restricción a un individuo (value)
Restricciones cardinales
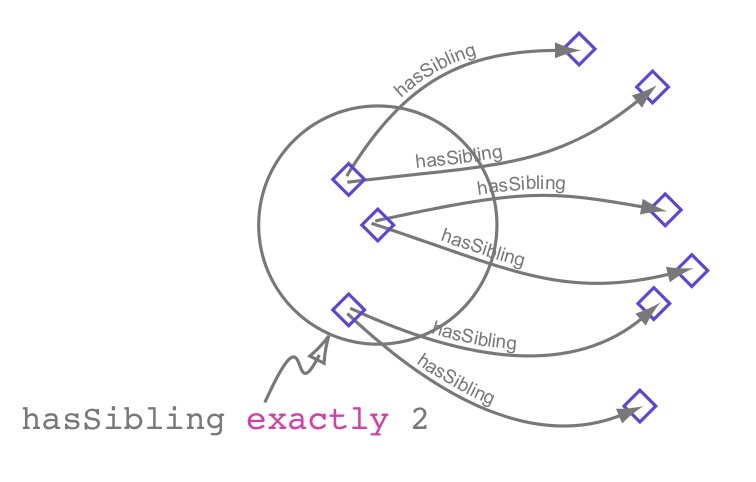
(+ QCR!) Manchester tutorial
Más axiomas para clases
disjointFrom
booleanos: not, or, and
Expresiones complejas
Propiedades OWL
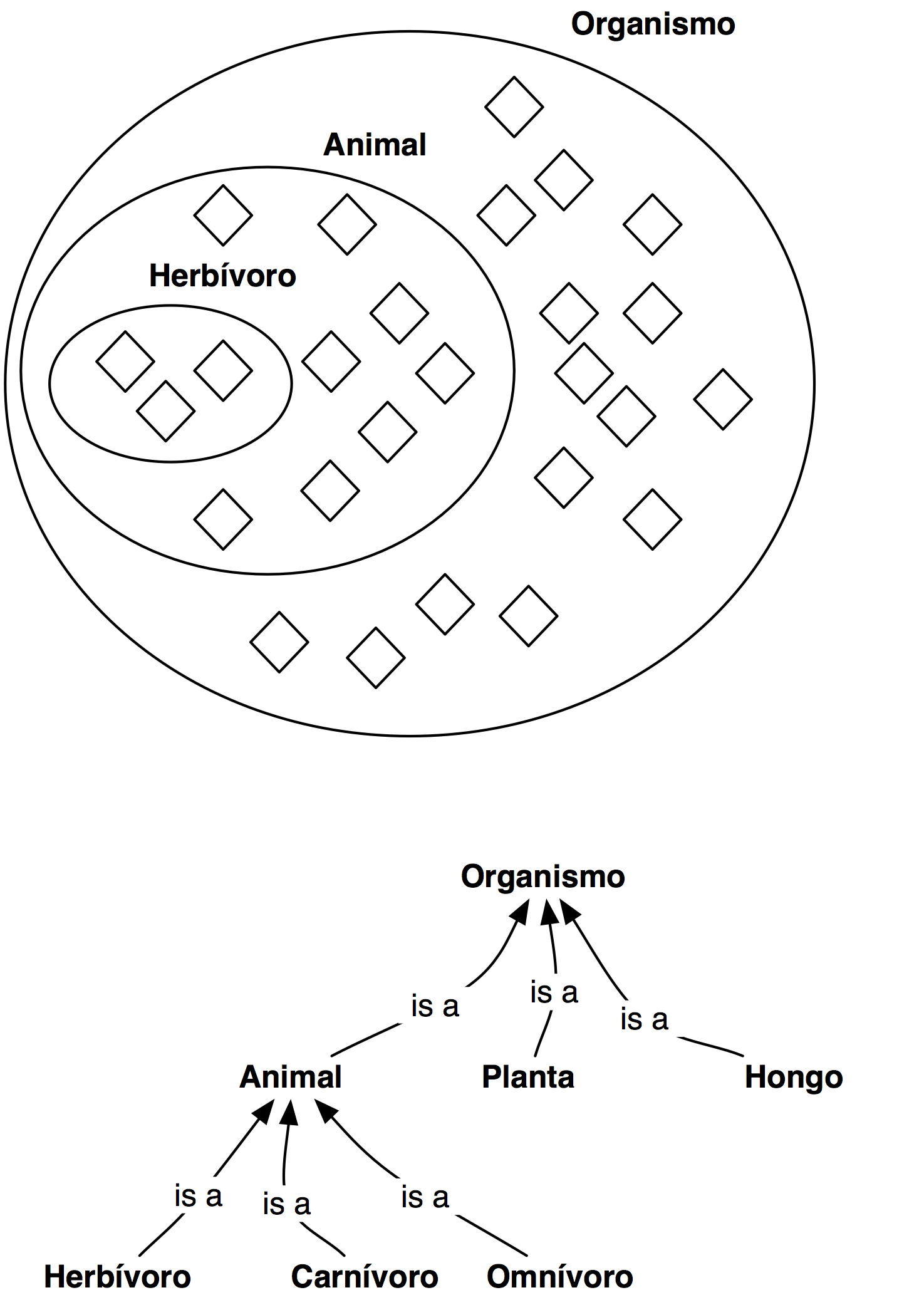
Jerarquía propiedades
Jerarquía propiedad-subpropiedad (~taxonomía pero con propiedades), ej:
- interacciona con
- mata a
- estrangula a
- mata a
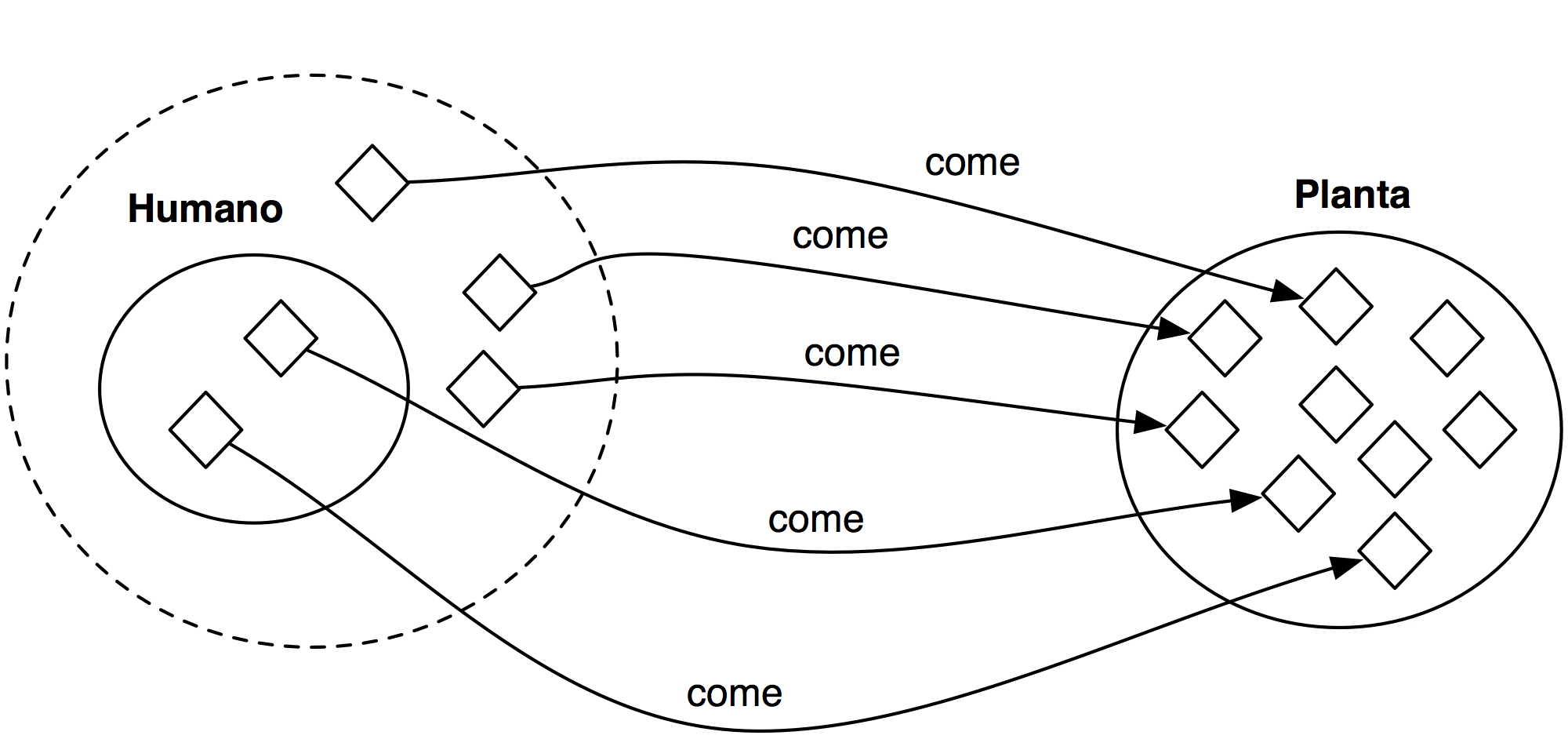
Características propiedades objeto
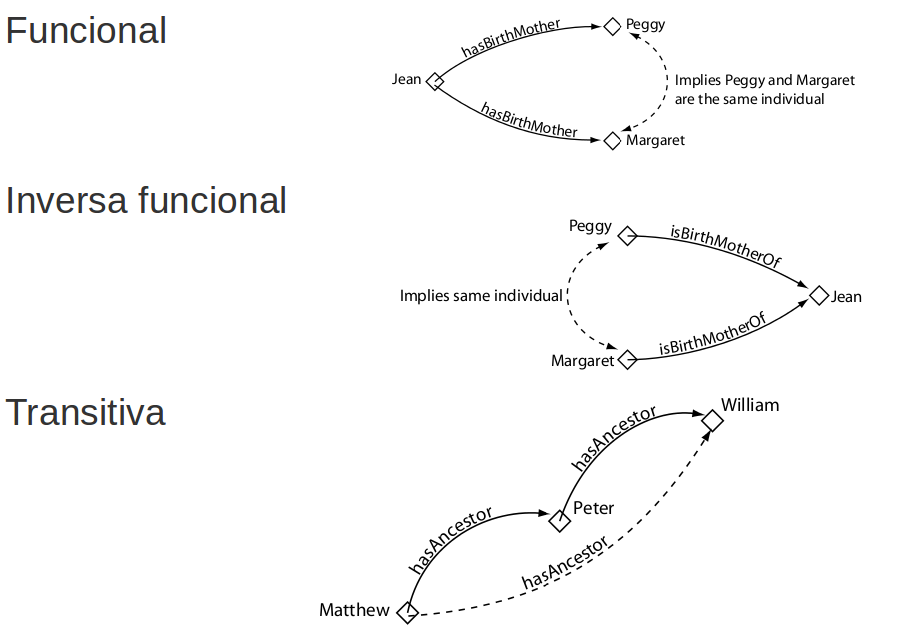
Características propiedades objeto
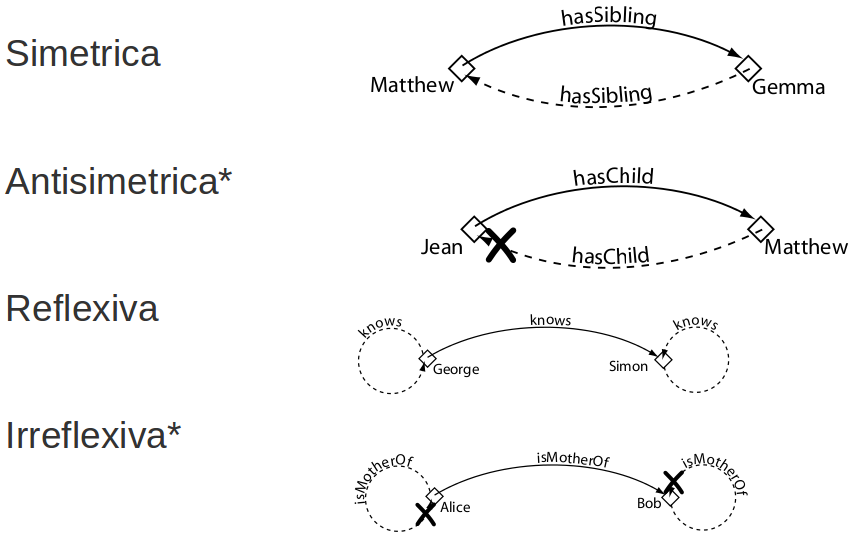
Características propiedades objeto
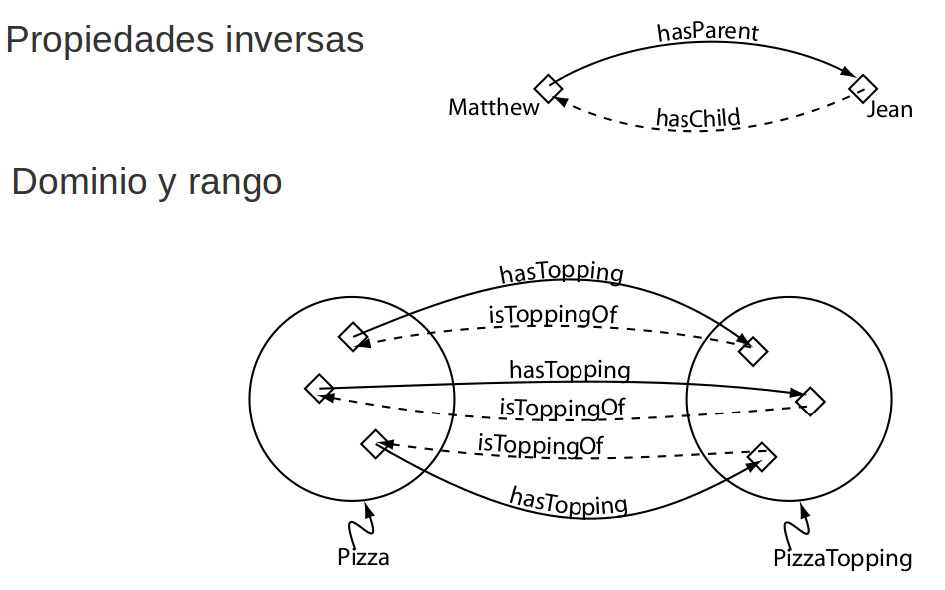
Características propiedades datos
Solo funcional
Dominio clases, rango datatypes
Propiedades anotación
Anotar con lenguaje natural entidades (propiedades, clases, individuos), axiomas, ontologías
Fuera de la semántica
rdfs:label, rdfs:comment, dublin core, a medida
Individuos
Miembro de una o más clases (Types)
Igual (SameAs) o diferente (DifferentFrom) a otro individuo
Relaciones binarias con otros individuos o datos (triples), positivas o negativas
Razonamiento automático
Un razonador infiere los "nuevos" axiomas que implican los axiomas que hemos introducido en la ontología
El razonador infiere todos los axiomas; es útil para tratar con conocimiento complejo
Open World Assumption (OWA)
(Falta de) Unique Name Assumption (¡owl:sameAs!)
Tareas más comunes razonamiento automático
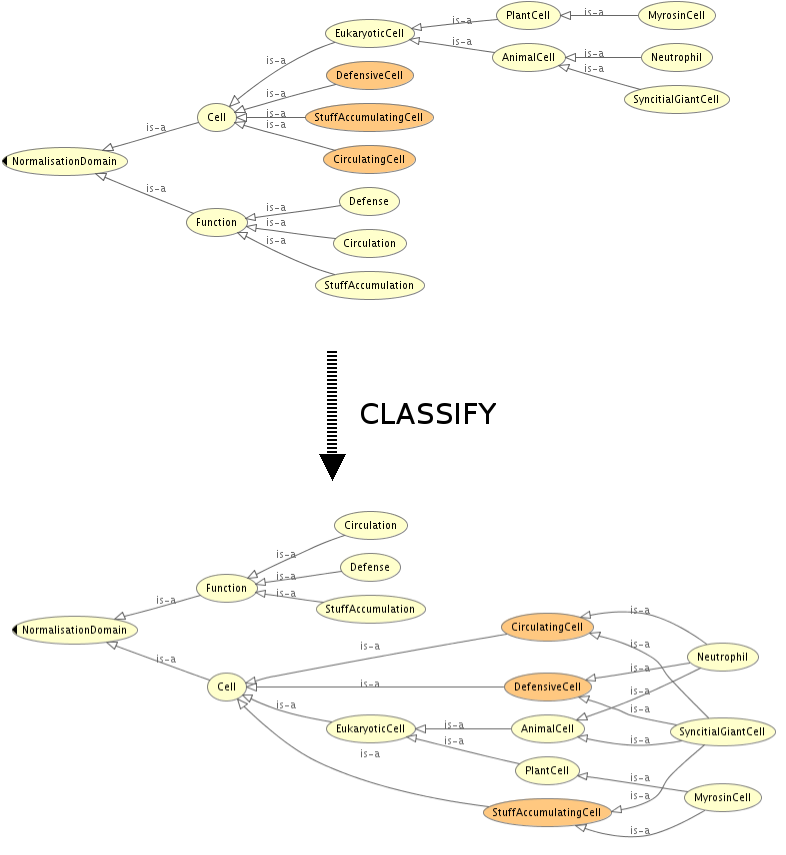
Mantener taxonomía
Consistencia
Clasificar entidades y consultas
Mantener una taxonomía
Consistencia
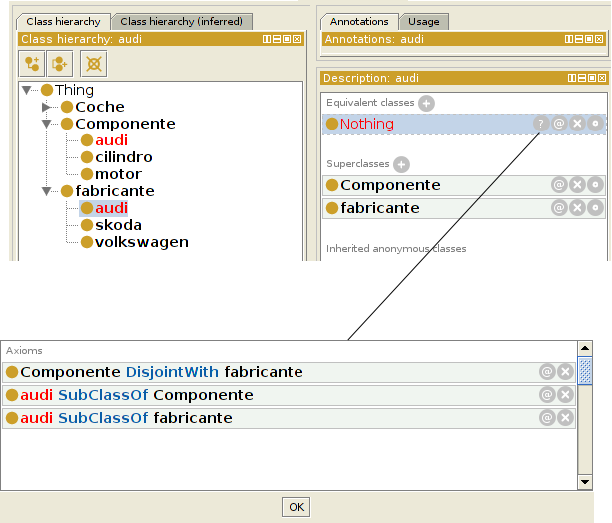
Clasificar entidades, consultas
Clasificar entidades: dada una entidad nueva, como se relaciona con las demas entidades (types, equivalentTo, subClassOf, triples)
Una consulta es una clase anónima que clasificamos contra la ontología como si fuese una entidad
Ejercicios
Ejercicios: owl_assignment.pdf
Soluciones:
SPARQL
SPARQL
Lenguaje para hacer consultas sobre grafos RDF (~"El SQL para RDF")
http://www.w3.org/standards/techs/sparql
SPARQL
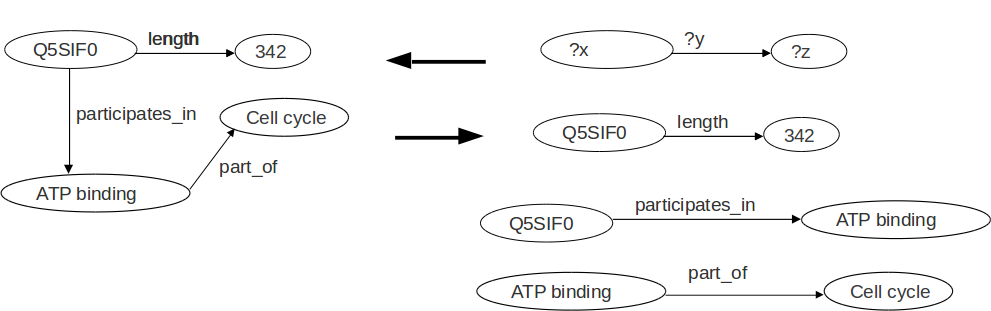
SPARQL
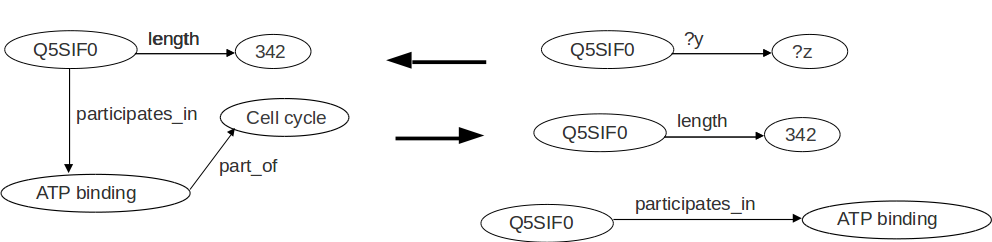
SPARQL
Tutorial: SPARQL by example (Cambridge Semantics)
SPARQL
Ejercicio práctico: http://linkedlifedata.com/sparql
Ejercicio práctico: BioGateway
SPARQL
Ejercicio práctico: http://miuras.inf.um.es/sparql
search for the human genetic diseases related to the gene Brca1 of the Mus musculus species stored in bio2rdf.org datasets
SPARQL
PREFIX ogolod: <http://miuras.inf.um.es/ogolod/ontology/>
SELECT DISTINCT ?bio2rdf_omim ?title
WHERE {
?gene owl:sameAs <http://bio2rdf.org/page/geneid:12189> .
?cluster ogolod:hasOrtholog ?gene .
?cluster ogolod:hasOrtholog ?ortholog .
?disease ogolod:causedBy ?ortholog .
?disease owl:sameAs ?bio2rdf_omim .
?disease ogolod:Name ?title .
}
SPARQL
Ejercicio práctico: http://miuras.inf.um.es/sparql
orthologs related to genes involved in lung cancer and belonging to Rattus Norvegicus
SPARQL
PREFIX ogolod: <http://miuras.inf.um.es/ogolod/ontology/>
PREFIX ogolodr: <http://miuras.inf.um.es/ogolod/resource/>
SELECT DISTINCT ?gene2
WHERE {
?ortholog ogolod:fromSpecies <http://miuras.inf.um.es/ogolod/resource/NCBITaxon_10116/NCBITaxon_10116> .
?ortholog owl:sameAs ?gene2 .
?cluster ogolod:hasOrtholog ?gene .
?cluster ogolod:hasOrtholog ?ortholog .
?disease ogolod:causedBy ?gene .
?disease owl:sameAs <http://bio2rdf.org/page/omim:211980> .
}
Cómo funciona LD
Grafos y triple stores
Un "triple store" contiene diferentes grafos
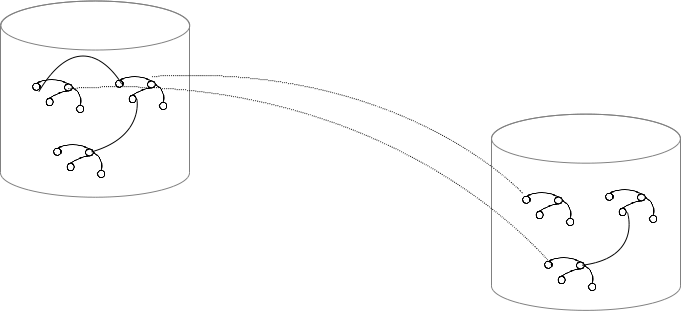
Consultas
Solo recibiremos los triples de ese triple store (¡Pero nosotros o nuestro agente automático podemos seguir los enlaces! "Follow your nose")
Alternativa: federated queries (http://www.w3.org/TR/sparql11-federated-query/)
Negociacion contenido
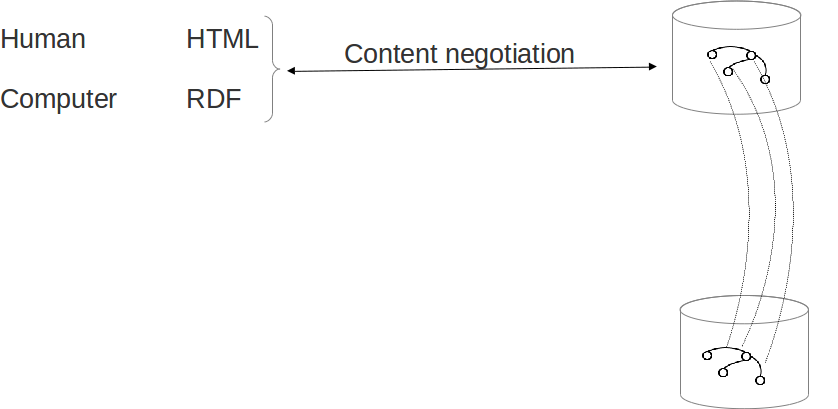
Negociacion contenido
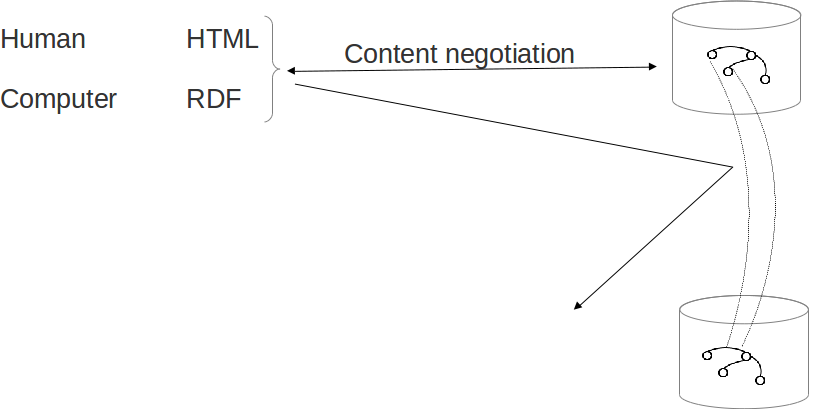
Negociacion contenido
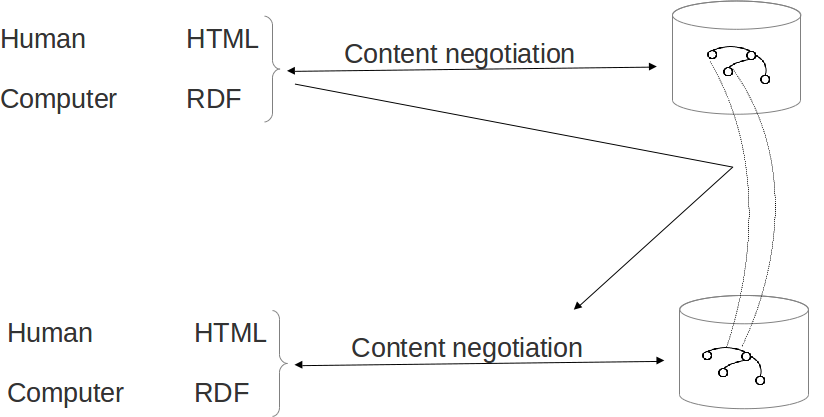
Diseño URIs
HTTP URIs dereferenciables: cuando se busca una URI, deberia devolver una descripción del objeto que identifica esa URI
Descripción del objeto (documento RDF) ≠ objeto
Diseño URIs
- Base URI: http://geo.linkeddata.es/
- TBox URIs:
- http://geo.linkeddata.es/ontology/{concept|property}
- http://geo.linkeddata.es/ontology/Provincia
- ABox URIs:
- http://geo.linkeddata.es/resource/{r. type}/{r. name}
- http://geo.linkeddata.es/resource/Provincia/Madrid
Inferencia en LD
Para producir el dataset: materializar triples y asegurar consistencia
En consultas
Consumir LD
Navegadores LD
Buscadores LD
Aplicaciones (Mash-ups)
- Revyu
- Talis aspire
- NYT
- ...
Publicar datos en LD
¿Por qué publicar datos en LD?
- Enlaces al exterior:
- Publicar solo nuestros datos, referancias al resto, no hay que replicar datos externos:
- Los datos externos se actualizan independientemente, y nuestro dataset va "a remolque" sin esfuerzo
¿Por qué publicar datos en LD?
- Enlaces a nuestro dataset:
- Es facil enlazar a nuestro dataset, ya que usamos HTTP URIs
- Por lo tanto, aumenta la capacidad de nuestro dataset de ser descubierto mediante enlaces
¿Por qué publicar datos en LD?
Semántica: el significado de nuestro datos es explícito y claro, debido a RDF (instancias) + OWL ("esquema"): es fácil crear aplicaciones, incluyendo razonamiento automático (ej. agentes)
Publicar datos en LD
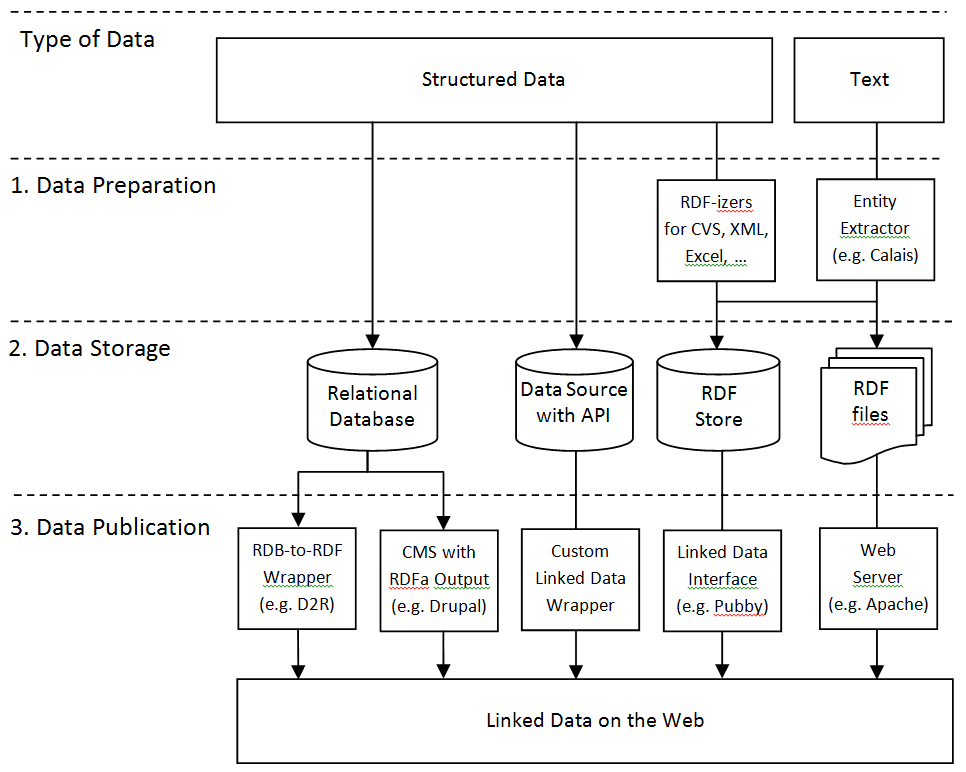
Publicar datos en LD
- Crear el dataset
- Ontología OWL: reusar lo más posible de otras ontologías para interoperabilidad
- Instancias RDF
- Añadir enlaces a otros datasets
- Manualmente o con herramientas como SILK
- A nivel de instancias (owl:sameAs, predicados, ...) y a nivel de vocabulario (owl:equivalentClass, ...)
Publicar datos en LD
- Almacenar el dataset en triple store
- Publicar el dataset mediante servidor web
- Registrar el dataset en Data Hub
- Generar archivo voiD (Vocabulary of Interlinked Datasets): http://www.w3.org/TR/void/
- Generar archivo sitemap.xml (con sitemap4RDF) y enviarlo a Semantic Web index (http://sindice.com/) y Google
Publicar datos en LD
Ejercicio práctico: recrear todo el proceso de publicar un dataset Linked Data
Crear el dataset y "publicarlo" en una infraestructura ya lista (Life Sciences Linked Data)
Life Sciences Linked Data
"Pack" ya listo y configurado para publicar Linked Data en localhost (Solo para GNU/Linux)
Life Sciences Linked Data
Fuseki: triple store
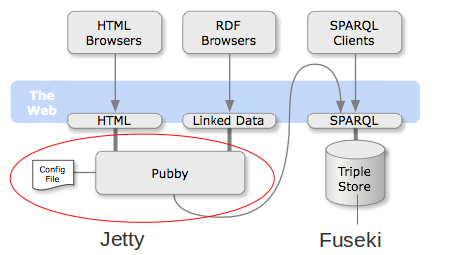
Jetty: servidor web
Pubby: negociacion contenido etc.
Life Sciences Linked Data
Probar con el dataset por defecto
Instrucciones: life-sciences-linked-data/LSLD/README, "Standard running"
Life Sciences Linked Data
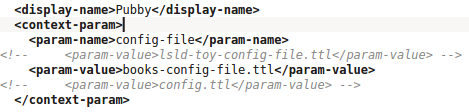
/jena-fuseki-0.2.6-SNAPSHOTchmod +x fuseki-server s-*./fuseki-server --update --mem /ds./s-put http://localhost:3030/ds/data default Data/books.ttl
Life Sciences Linked Data
jetty-distribution-9.0.0.M3/webapps/ROOT/WEB-INF/web.xml/jetty-distribution-9.0.0.M3java -jar start.jar jetty.port=8080- http://localhost:8080/book1
Life Sciences Linked Data
Publicar el dataset de ejemplo con enlaces a otros datasets LOD
Instrucciones: life-sciences-linked-data/LSLD/README, "Using a different dataset"
Life Sciences Linked Data
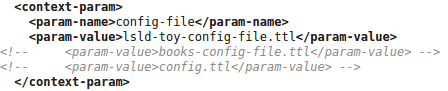
/jena-fuseki-0.2.6-SNAPSHOTmkdir lslddb./fuseki-server --update --loc=./lslddb /dataset./s-put http://localhost:3030/dataset/data lsld Data/LSLD_example.owl
Life Sciences Linked Data
jetty-distribution-9.0.0.M3/webapps/ROOT/WEB-INF/web.xml/jetty-distribution-9.0.0.M3java -jar start.jar jetty.port=8080- http://localhost:8080/Protein_A
Life Sciences Linked Data
Crear vuestro propio dataset con Protégé 4 (http://protege.stanford.edu/) (Demo Protégé?)
Con enlaces a otros datasets
Publicar el dataset en localhost (Usar "Using a different dataset" como guía)
Life Sciences Linked Data
Silk ...
Inferencia con P4: materializar triples ...
Programación para LD
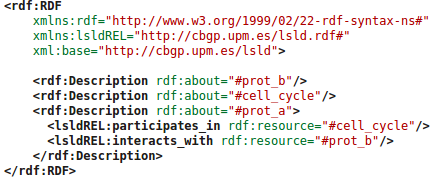
Generar RDF con Jena
Model model = ModelFactory.createDefaultModel();
Resource prot_a = model.createResource("http://cbgp.upm.es/lsld.rdf#prot_a");
Resource cell_cycle = model.createResource("http://cbgp.upm.es/lsld.rdf#cell_cycle");
Property participates_in = model.createProperty("http://cbgp.upm.es/lsld.rdf#participates_in");
model.add(prot_a, participates_in,cell_cycle);
model.write(System.out);
Aplicaciones Linked Data
Consulta SPARQL endpoint con JENA (Juan F. Sequeda)
import com.hp.hpl.jena.query.*;
String service = "..."; // address of the SPARQL endpoint
String query = "SELECT ..."; // your SPARQL query
QueryExecution e = QueryExecutionFactory.sparqlService(service, query)
ResultSet results = e.execSelect();
while ( results.hasNext() ) {
QuerySolution s = results.nextSolution();
// ...
}
e.close();
Sumario herramientas para LD
- Triple strores y APIs: Jena, Virtuoso, Sesame, OWL API, ...
- Editores ontologías: Protégé, TopBraid composer, ...
- Publicar LD: Silk, Pubby, ...
- Validadores: Vapour, RDF:Alerts, Sindice inspector, ...
- ...
Más información, agradecimientos, etc.
Más información
Linked Data, the story so far (Christian Bizer, Tom Heath, Tim Berners-Lee)
Linked Data: Evolving the Web into a Global Data Space (Christian Bizer, Tom Heath)
Semantic Web Health Care and Life Sciences Interest Group (W3C HCLS IG): Health Care and Life Science (HCLS) Linked Data Guide
Más información
José Antonio Miñarro-Giménez, Mikel Egaña Aranguren, Boris Villazón-Terrazas and Jesualdo Tomás Fernández-Breis. Publishing Orthology and Diseases Information in the Linked Open Data cloud. Current Bioinformatics 2012, 7 (3), 255-266. [Bentham science] [pdf]
Inspiración
- Ontology engineering Group (UPM): Raúl García Castro
- Bioinformatics and Semantic Web Group, University of Texas: Juan F. Sequeda
- Isoco: Boris Villazón Terrazas
- Biological Informatics Group (CBGP, UPM): Mark Wilkinson
- Linked Data: Evolving the Web into a Global Data Space (Christian Bizer, Tom Heath)