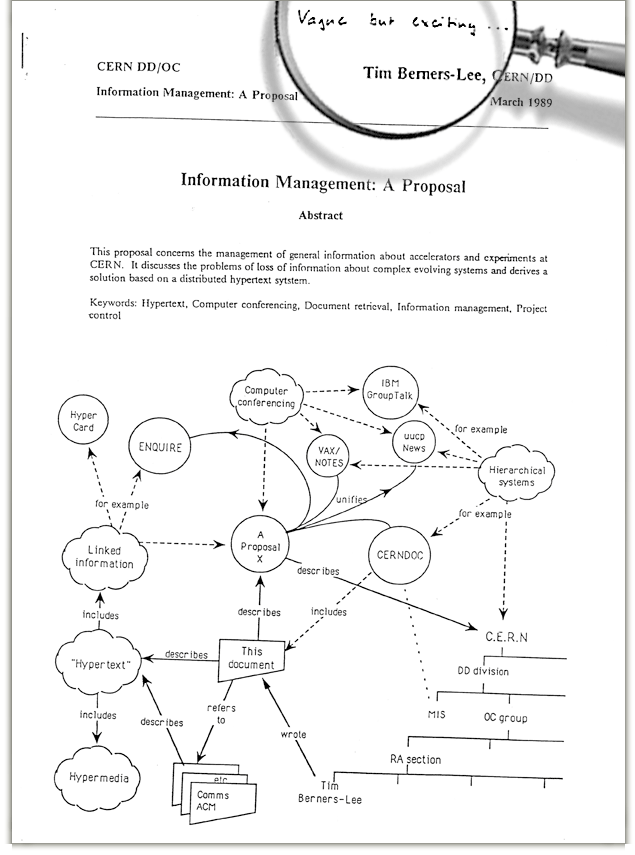
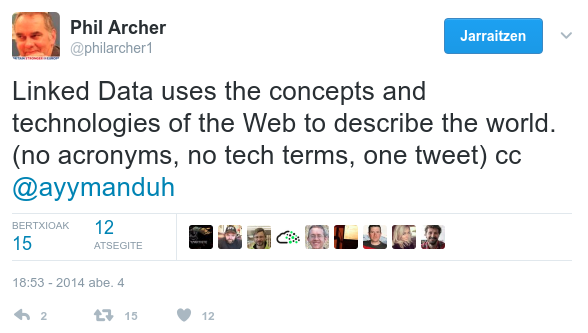
Life Sciences Semantic Web
Máster Bioinformática UM 2016 - 2017

Life Sciences Semantic Web

Web - 1990s
Web - 2010s



Web Semántica

Linked Data
Linked Data
Principios Linked Data
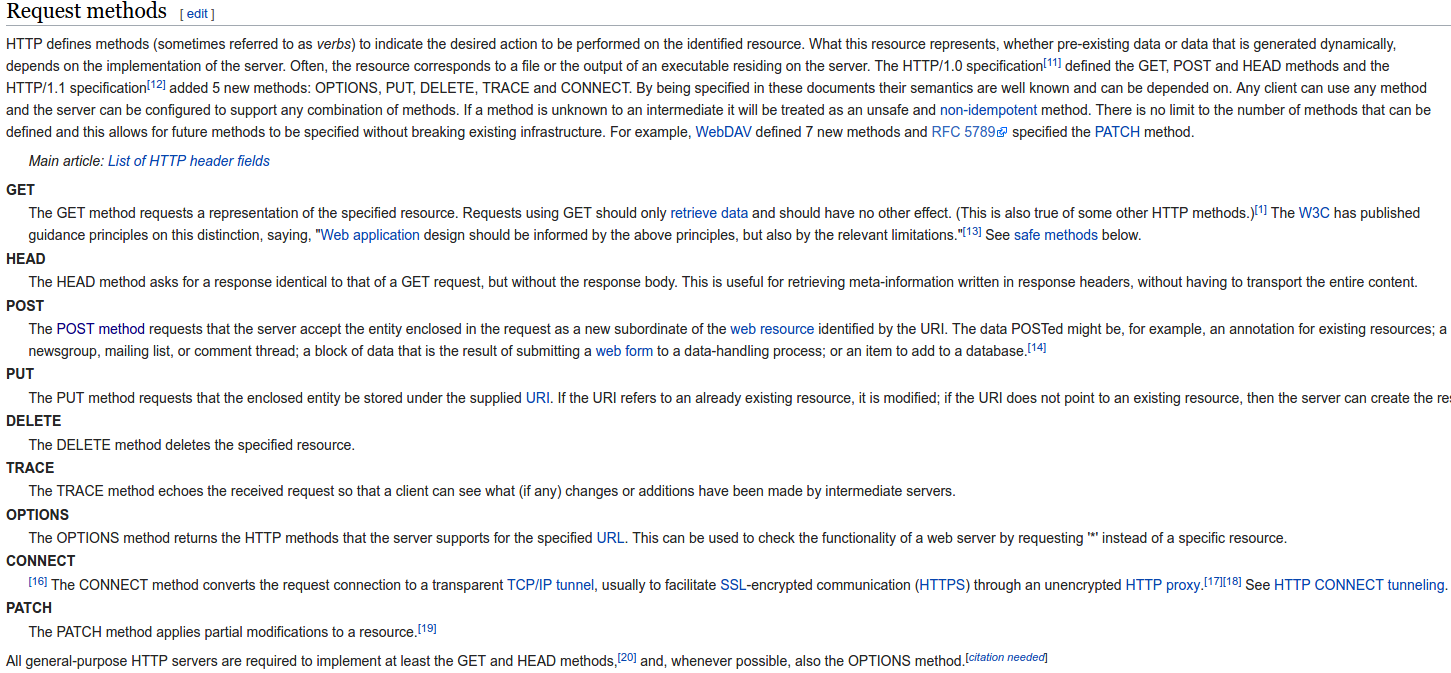
1.- Usar URIs (Uniform Resource Identifier) para identificar entidades
2.- Usar URIs que son accesibles mediante el protocolo HTTP, para que usuarios o agentes automáticos puedan acceder a las entidades
3.- Cuando se acceda a la entidad, proveer datos sobre la entidad en formatos estándar y abiertos, como RDF (Resource Description Framework)
4.- Añadir en los datos que publicamos en RDF enlaces a las URIs de otras entidades, de modo que un usuario o agente pueda navegar por la red de datos y descubrir más datos que también siguen los principios Linked Data

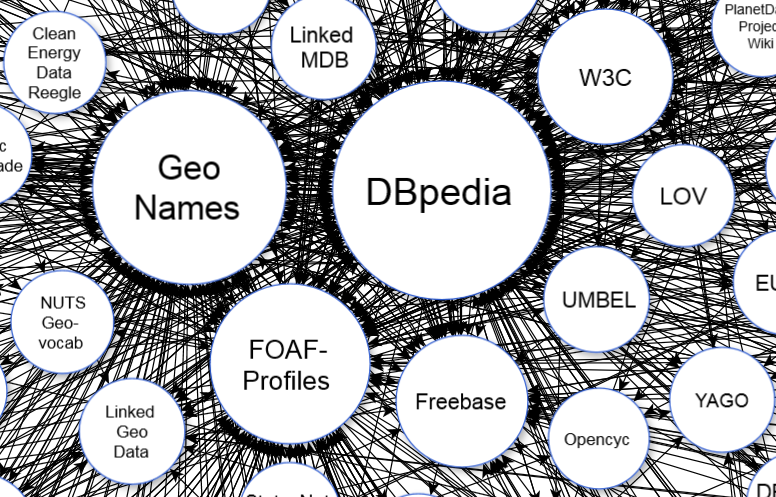
Linked Open Data cloud

Linked Open Data cloud 2014
Linked Open Data cloud 2017
Ontologías (ej. Gene Ontology)
Datos Enlazados (Linked Data)
Vocabulario de anotación
Razonamiento automático
Life Sciences Semantic Web
W3C Semantic Web stack
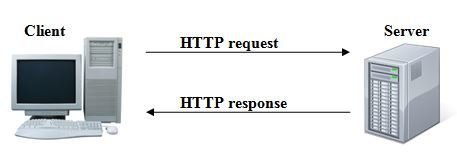
Arquitectura Web


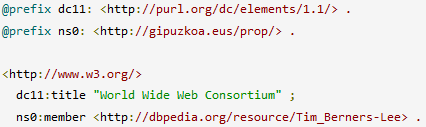
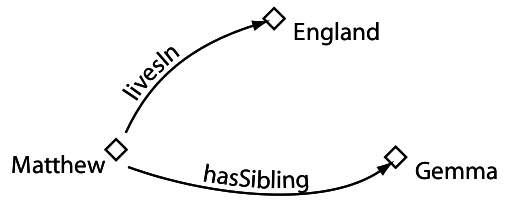
RDF
RDF (Resource Description Framework)
Triple RDF
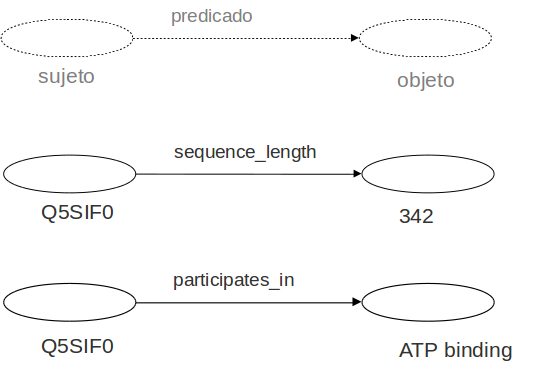
Grafo RDF
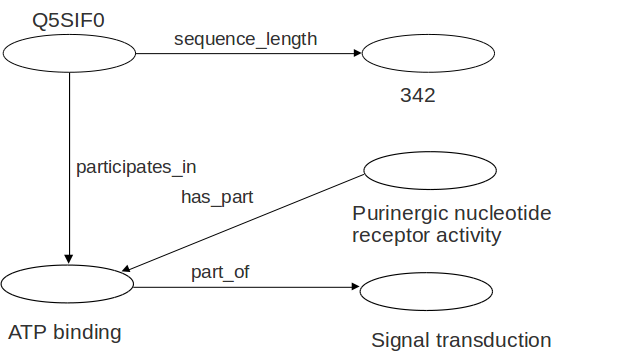
Grafo RDF
Todas las entidades del grafo se identifican mediante URIs
URI: Uniform Resource Identifier (≠ URL!). Identifica recursos
URL: Uniform Resource Locator. Una URI que indica la localización física de un recurso en la red

Grafo RDF: URIs
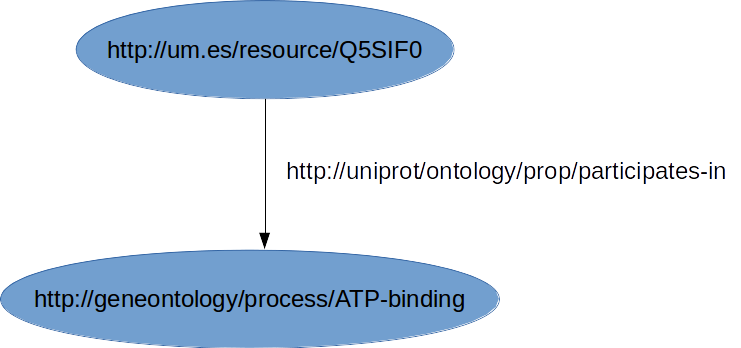
Grafo RDF
Los sujetos y predicados sólo pueden ser recursos (URIs)
Algunos objetos pueden ser valores literales (Cadenas de caracteres)
Los valores literales pueden tener tipo (XML Schema datatypes)
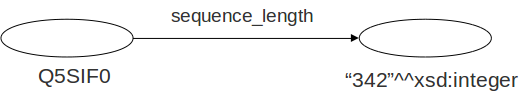
rdf:type
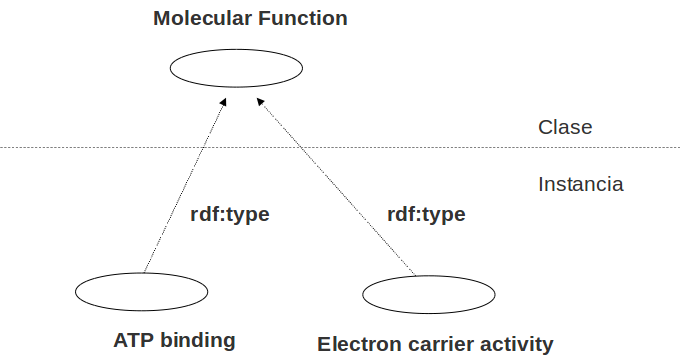
Agrupar recursos en clases
RDF namespaces
RDF usa namespaces para "agrupar" URIs
Namespaces se pueden abreviar/expandir mediante prefix
PREFIX dbpedia:<http://dbpedia.org/resource/>
dbpedia:Donostia = http://dbpedia.org/resource/Donostia
dbpedia:Ataun = http://dbpedia.org/resource/Ataun
...
Vocabularios
RDF |
http://www.w3.org/1999/02/22-rdf-syntax-ns# |
RDFS |
http://www.w3.org/2000/01/rdf-schema# |
OWL |
http://www.w3.org/2002/07/owl# |
Vocabulario: informalmente, colección definida de URIs, normalmente bajo un mismo namespace
Vocabularios "reservados" (definen lenguajes)
rdf:type = http://www.w3.org/1999/02/22-rdf-syntax-ns#type
Ontologías
La mayoría de los vocabularios son ontologías
Definen propiedades generales de los datos que queremos publicar:
foaf:person dbpedia-ont:city dc:book ...
Ontologías
Prefix.cc
Linked Open Vocabularies
BioPortal
Ontobee
RDF: modelo vs sintaxis
RDF es un modelo de datos
Ese modelo abstracto se puede representar con diferentes sintaxis: "Serializar" (escribir) en un archivo
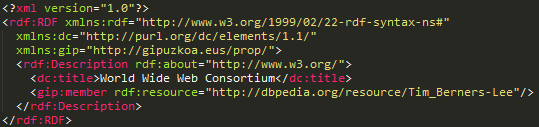
Una de esas sintaxis es RDF/XML
No confundir el modelo con la sintaxis: ¡RDF es mucho más que un archivo XML!
Serializar RDF
RDF/XML (http://www.w3.org/TR/rdf-syntax-grammar/)
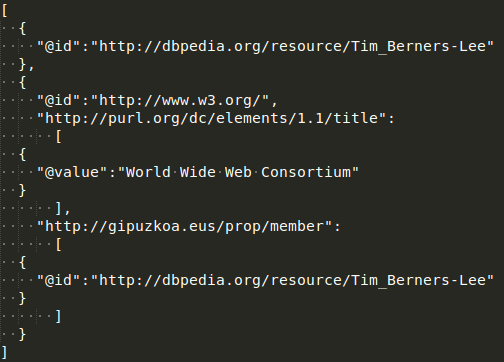
JSON-LD (https://www.w3.org/TR/json-ld/)
RDFa (http://www.w3.org/TR/rdfa-core/)
Turtle (http://www.w3.org/TR/turtle/)
Serializar RDF: RDF/XML

Serializar RDF: JSON-LD

Serializar RDF: Turtle

Herramientas utiles
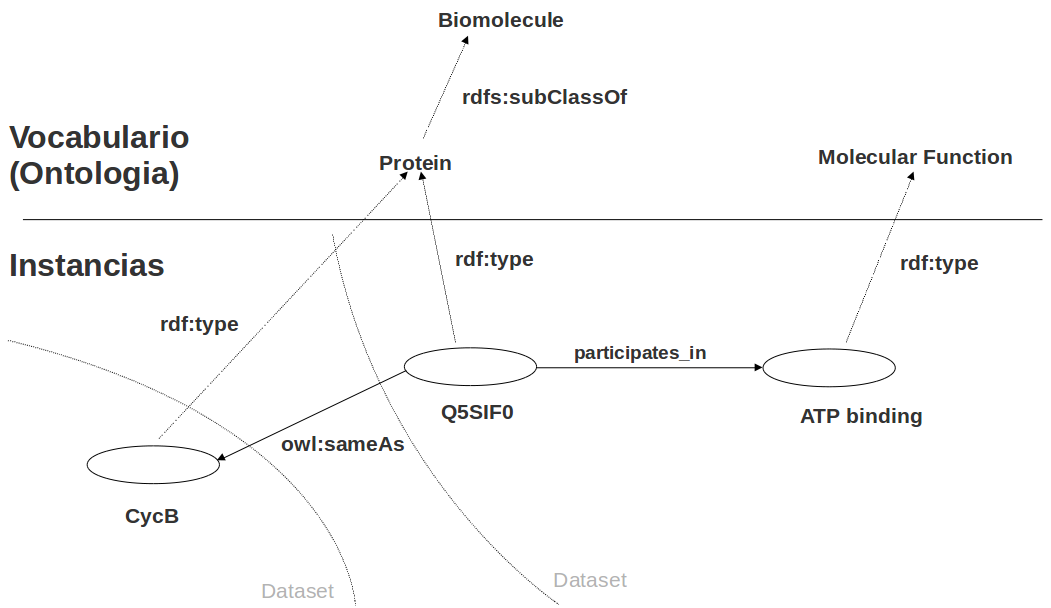
Ejercicio
Escribir este modelo RDF en un archivo turtle
Ejercicio
1) Texto plano (TTL)
2) Convertir de turtle a RDF/XML (http://www.easyrdf.org/converter)
3) Visualizar en validador W3C
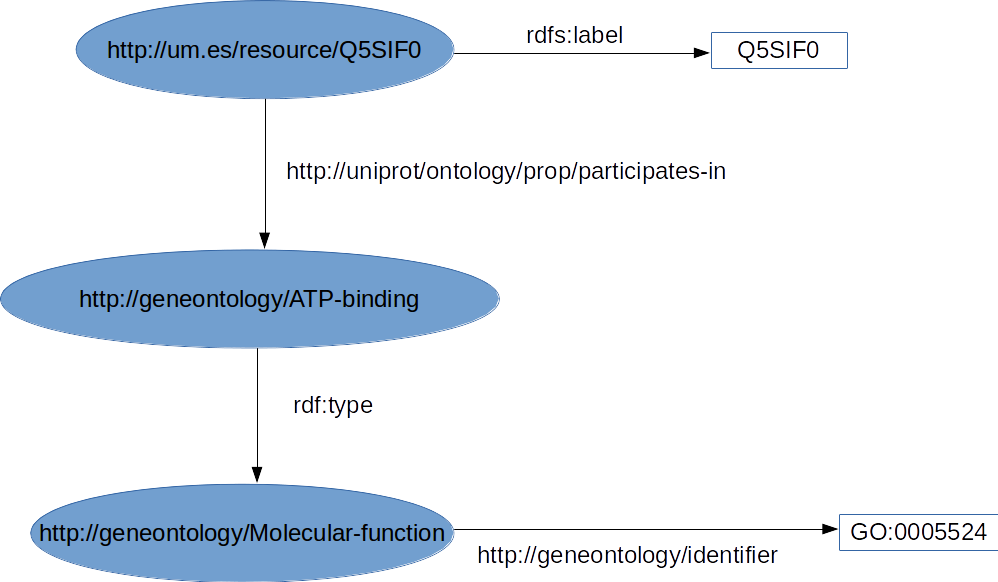
Ejercicio
Prefixes:
um: <http://um.es/resource/>
uniprot: <http://uniprot/ontology/prop/>
go: <http://geneontology/>
rdf: ????
rdfs: ????
Ejemplo:
Solución

SPARQL
SPARQL
Lenguaje para hacer consultas sobre grafos RDF (~"El SQL para RDF")
Triple Store
~# ssh user@biomaster.atica.um.es ~# /home/user
~# git clone https://github.com/mikel-egana-aranguren/LSSW-UM-2016-2017 ~# cd LSSW-UM-2016-2017/LinkedDataServer/blazegraph
~# java -server -Djetty.port=8081 -jar bigdata-bundled.jar
[Subir RDF/data.rdf y RDF/data2.rdf]
Familiarizarse con RDF ejemplo
Estructura de consulta
[Ejecutar ejemplos en Triple Store]
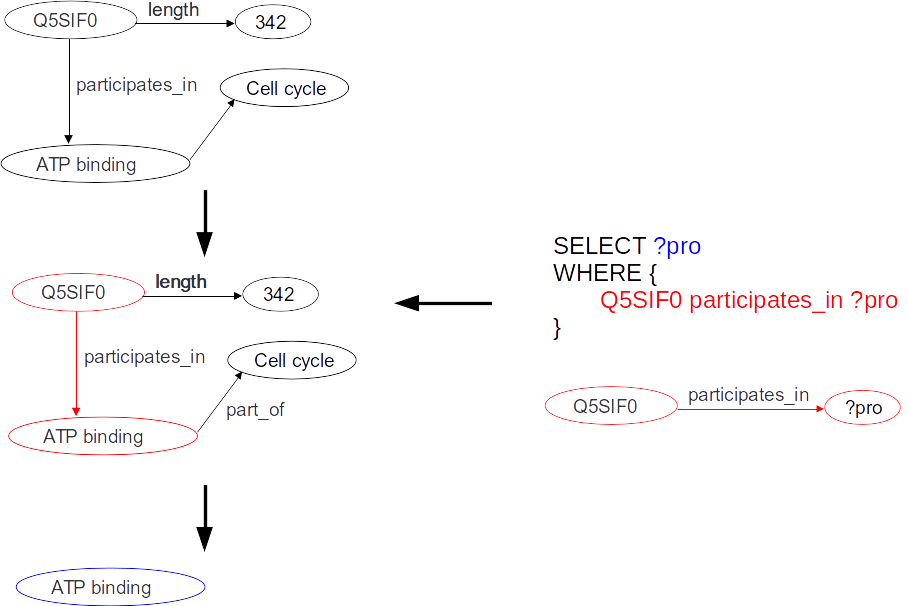
# Variables que queremos recibir SELECT ?s ?p ?o # Patrón del grafo que queremos extraer del grafo mayor WHERE { ?s ?p ?o }
Estructura de consulta
# Prefixes PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> # Variables que queremos recibir SELECT ?p # Patrón del grafo que queremos extraer del grafo mayor WHERE { ?p gr_ont:part_of gr_data:Nucleolus }
Estructura de consulta
# Prefixes PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> # Variables que queremos recibir SELECT ?p ?clase # Patrón del grafo que queremos extraer del grafo mayor WHERE { ?p gr_ont:part_of gr_data:Nucleolus . ?p rdf:type ?clase }
Optional
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> SELECT ?p ?label ?part WHERE{ # Tiene que ser parte de algo ?p gr_ont:part_of ?part . # Tiene que tener un rdfs:label ?p rdfs:label ?label }
Optional
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> SELECT ?p ?label ?part WHERE{ # Tiene que ser parte de algo ?p gr_ont:part_of ?part . # Tiene que tener un rdfs:label, o no OPTIONAL{ ?p rdfs:label ?label } }
Union
# Grafos alternativos PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> SELECT ?name WHERE{ {?p gr_ont:name ?name} UNION {?p rdfs:label ?name} }
Filtrar resultados
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> SELECT ?name WHERE{ {?p gr_ont:name ?name} UNION {?p rdfs:label ?name} FILTER regex(?name,'Protein.*') }
Filtrar resultados
Logica: !, &&, ||
Calculos: +, -, *, /
Comparaciones: =, !=, >,<
Tests SPARQL: isURI, isBlank, isLiteral, bound
Acceder a datos: str, lang, datatype
Más: sameTerm, langMatches, regex, ...
ASK
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> ASK WHERE{ gr_data:Gene_B gr_ont:name "Gene B" }
DESCRIBE
DESCRIBE <http://genomic-resources.eu/resource/Prot_B>
CONSTRUCT
PREFIX gr_ont: <http://genomic-resources.eu/ontology/>
PREFIX gr_data: <http://genomic-resources.eu/resource/>
PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#>
PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#>
CONSTRUCT {
?p rdf:type gr_data:NucleolusPart .
gr_data:NucleolusPart rdfs:label "Part of the nucleolus"
}
WHERE {
?p gr_ont:part_of gr_data:Nucleolus
}
INSERT
[Pestaña "Update" en Blazegraph, seleccionar "SPARQL update"]
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#> INSERT { GRAPH <http://genomic-resources.eus/graph/um>{ ?p rdf:type gr_data:NucleolusPart . gr_data:NucleolusPart rdfs:label "Part of the nucleolus" } } WHERE { ?p gr_ont:part_of gr_data:Nucleolus }
GRAFOS
Grafo: conjunto de triples
El conjunto entero se identificada con una URI (diferente de la de los datos)
Todas las Triple Stores tienen un Default Graph
GRAFOS

GRAFOS
Los grafos son muy útiles para añadir datos sobre los datos: ej. procedencia, autoría, fecha de generación
(entre otras cosas)
GRAFOS
SELECT *
FROM <http://genomic-resources.eus/graph/um>
WHERE{
?s ?p ?o
}
SELECT *
FROM <http://genomic-resources.eus/graph/um>
WHERE{
?s ?p ?o
}
INSERT DATA
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#> INSERT DATA { GRAPH <http://genomic-resources.eus/graph/um>{ gr_data:NucleolusPart rdfs:comment "Comment inserted" } }
DELETE DATA
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#> DELETE DATA { GRAPH <http://genomic-resources.eus/graph/um>{ gr_data:NucleolusPart rdfs:comment "Comment inserted" } }
DELETE
PREFIX gr_ont: <http://genomic-resources.eu/ontology/> PREFIX gr_data: <http://genomic-resources.eu/resource/> PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#> DELETE { GRAPH <http://genomic-resources.eus/graph/um>{ gr_data:NucleolusPart rdfs:label ?label } } WHERE { GRAPH <http://genomic-resources.eus/graph/um>{ gr_data:NucleolusPart rdfs:label ?label } }
Consultas federadas
PREFIX up:<http://purl.uniprot.org/core/>
SELECT ?prot ?protein
WHERE{
?prot rdfs:comment 'A4_HUMAN'
SERVICE <http://sparql.uniprot.org/>{
?protein a up:Protein .
?protein up:mnemonic 'A4_HUMAN'
}
}Ejemplos de Consultas (federadas)
UNIPROT
UNIPROT 1
¿Cuantas entradas de proteinas hay en uniprot?
https://github.com/mikel-egana-aranguren/LSSW-UM-2016-2017/tree/master/SPARQL/UniProt-1
UNIPROT 2
¿Cuantas entradas de proteinas hay en uniprot, que sean de Arabidopsis thaliana?
https://github.com/mikel-egana-aranguren/LSSW-UM-2016-2017/tree/master/SPARQL/UniProt-2
UNIPROT 3
¿Cuál es la descripción de la actividad enzimática de uniprotkb:Q9SZZ8?
https://github.com/mikel-egana-aranguren/LSSW-UM-2016-2017/tree/master/SPARQL/UniProt-3
UNIPROT 4
Conseguir los IDs, y las fechas en las que se añadieron a UniProt, de las proteinas que se han añadido este año a UniProt (Google: "SPARQL FILTER by date")
https://github.com/mikel-egana-aranguren/LSSW-UM-2016-2017/tree/master/SPARQL/UniProt-4
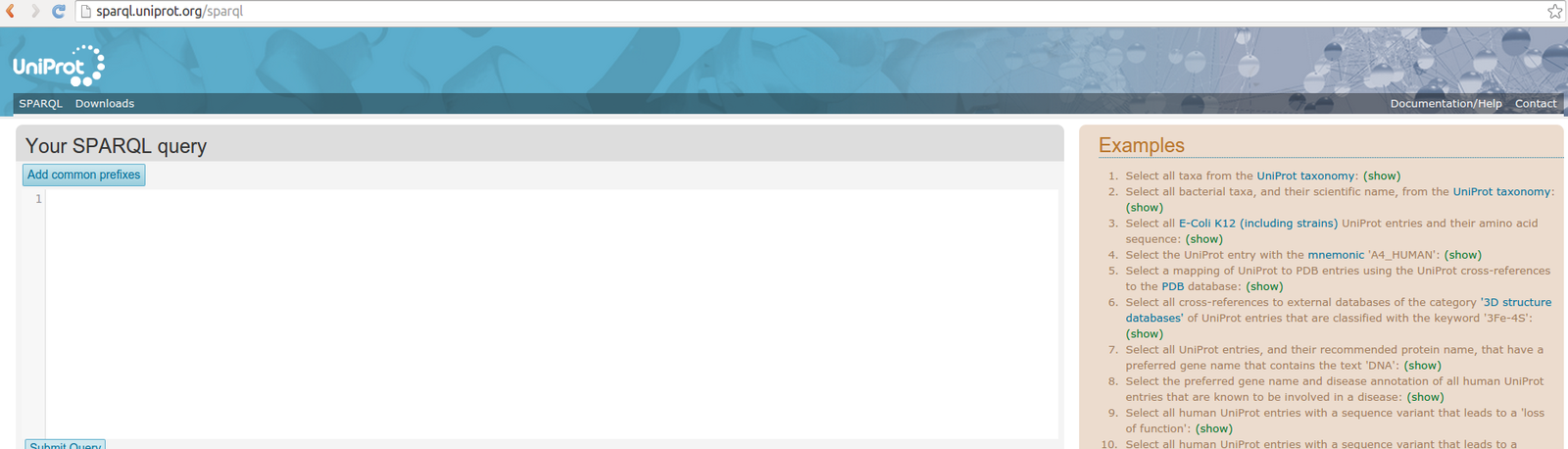
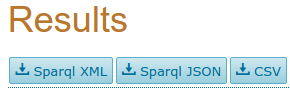
Cliente SPARQL
curl --data-urlencode query@UniProt-4 http://sparql.uniprot.org/sparql/
Resultados en XML, JSON, CSV?
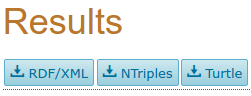
Cliente SPARQL
curl --data-urlencode query@describe http://sparql.uniprot.org/sparql/
DESCRIBE <http://purl.uniprot.org/uniprot/P05067>
Resultados en RDF/XML, Turtle?
Cliente SPARQL
Redirigir resultado describe a un archivo
Buscar en resultado describe "GO_0008201" y redirigir nuevo resultado a un archivo

OWL
OWL
OWL (Web Ontology Language) es un estándar oficial del W3C para crear ontologías en la web con un semántica precisa y formal
OWL
OWL se basa en Lógica Descriptiva (DL)
Representación computacional de un dominio de conocimiento:
Razonamiento automático: inferir conocimiento "nuevo" (*), consultas, consistencia, clasificar entidades contra la ontología, ...
Integrar conocimiento disperso
Sintaxis
Para ordenadores: RDF/XML, OWL/XML, ...
Para humanos: Manchester OWL Syntax, functional, ...


Semántica
Una ontología OWL esta compuesta de:
-
Entidades: las entidades del dominio de conocimiento, identificadas con URIs, introducidas por el desarrollador ("Mikel", "participa_en", ...)
-
Axiomas: relacionan las entidades mediante el vocabulario lógico que ofrece OWL (namespace OWL)
Una ontología puede importar otra (owl:import) y hacer referencia a sus entidades mediante axiomas
Entidades
Entidades (URI)
Axiomas
("URI OWL")
Individuos
Clases
Propiedades
Objeto
Datos
Anotación
Ontologia
(URI)
Entidades
Entidad
Axioma
Arm subClassOf part-of some Body
Clase
Clase
Propiedad objeto
Restricción
Individuos

[Fuente de imagenes: Manchester OWL Pizza tutorial]
Propiedades
Clases

Clases

Clase subclase

Clases equivalentes

Jerarquía de clases (Taxonomía)

Condiciones necesarias

Condiciones necesarias y suficientes

Restricción existencial

Restricción universal
Restricción un individuo (value)

Restricciones cardinales

Más axiomas
disjointFrom
booleanos: not, or, and

Expresiones complejas

Propiedades

Jerarquía Propiedades
Jerarquía propiedad-subpropiedad (~taxonomía pero con propiedades), ej:
-
interacciona con
-
mata a
estrangula a
-
Propiedades

Propiedades

Propiedades

Propiedades datos
Solo funcional
Dominio clases, rango datatypes
Propiedades anotación
Anotar con lenguaje natural entidades (propiedades, clases, individuos), axiomas, ontologías
Fuera de la semántica
rdfs:label, rdfs:comment, dublin core, a medida
Individuos
Miembro de una o más clases (Types)
Igual (SameAs) o diferente (DifferentFrom) a otro individuo
Relaciones binarias con otros individuos o datos (triples), positivas o negativas
Razonamiento automático
Un razonador infiere los "nuevos" axiomas que implican los axiomas que hemos introducido en la ontología
El razonador infiere todos los axiomas; es útil para tratar con conocimiento complejo
Open World Assumption (OWA)
(Falta de) Unique Name Assumption (¡owl:sameAs!)
Razonamiento automático
Mantener taxonomía

Razonamiento automático
Consistencia

Razonamiento automático
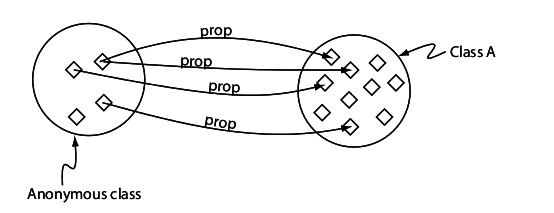
Clasificar entidades: dada una entidad nueva, como se relaciona con las demas entidades (types, equivalentTo, subClassOf, triples)
Una consulta es una clase anónima que clasificamos contra la ontología como si fuese una entidad
Ejemplo
Abrir rdf/ontology.owl
Importar rdf/data-ont.rdf
owl:ObjectProperty!!!
Explorar
Gene and part_of some Organelle
Algun otro ejemplo?

Linked Data
Principios Linked Data
1.- Usar URIs (Uniform Resource Identifier) para identificar entidades
2.- Usar URIs que son accesibles mediante el protocolo HTTP, para que usuarios o agentes automáticos puedan acceder a las entidades
3.- Cuando se acceda a la entidad, proveer datos sobre la entidad en formatos estándar y abiertos, como RDF (Resource Description Framework)
4.- Añadir en los datos que publicamos en RDF enlaces a las URIs de otras entidades, de modo que un usuario o agente pueda navegar por la red de datos y descubrir más datos que también siguen los principios Linked Data
Linked Data
Utilizar maquinaria Web (URIs HTTP), para identificar y localizar entidades: http://example.com/entity
Utilizar un modelo de datos común, tripleta RDF, para integrar datos en los que aparecen esa entidades

«base de datos universal»
Ventajas Linked Data
Descubrimiento e integración de datos
Programación de agentes que consuman los datos
Actualización de datos mediante enlaces
Consultas complejas
Ventajas Linked Data
Con Linked Data cualquiera puede publicar datos y enlazarlos a otros datos
El conjunto de datos abiertos publicados mediante Linked Data forma la «nube Linked Open Data»
Cada vez más instituciones públicas de todo el mundo usan Linked Data para publicar sus datos


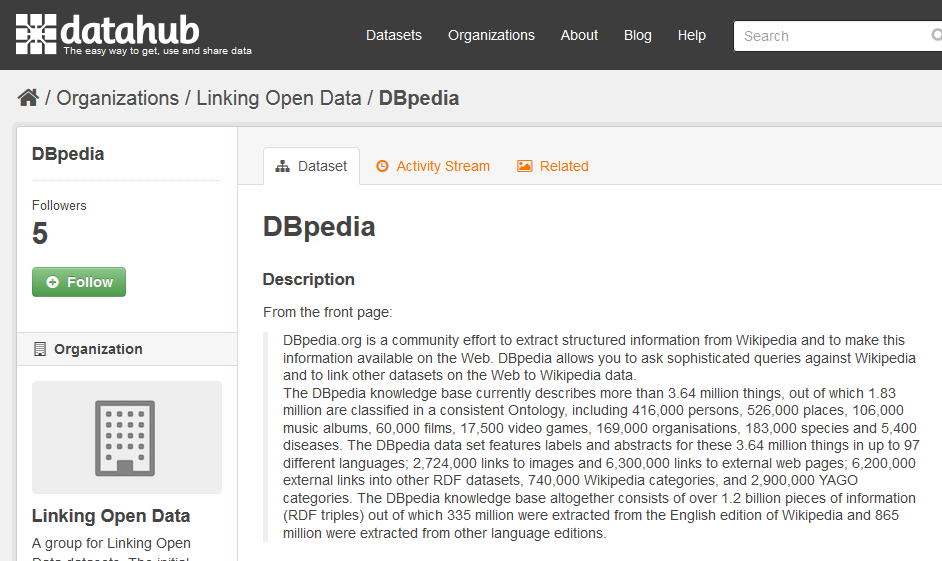
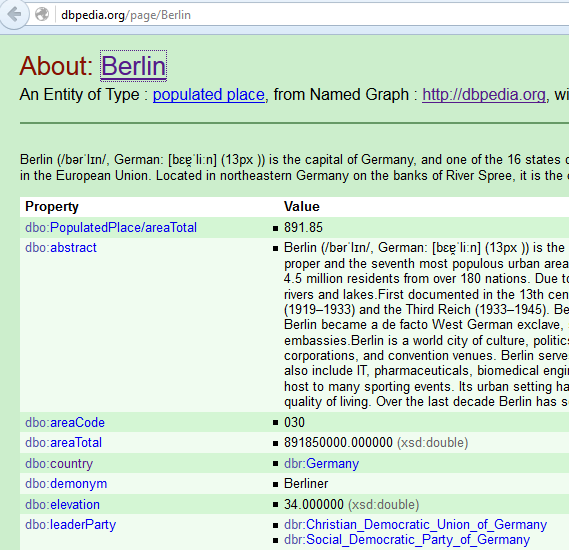
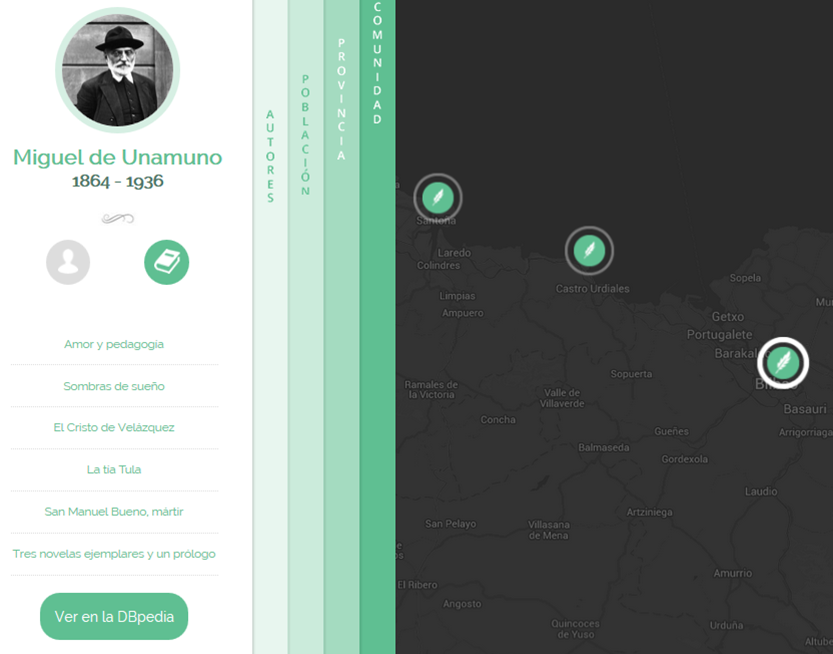
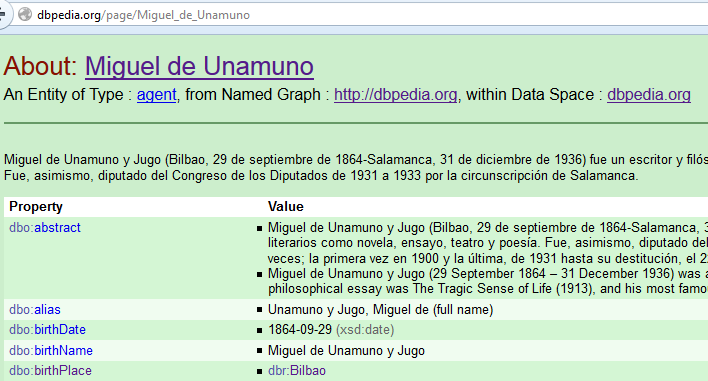
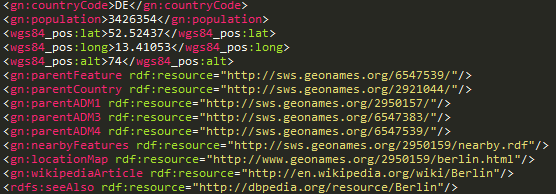
http://dbpedia.org/resource/Berlin
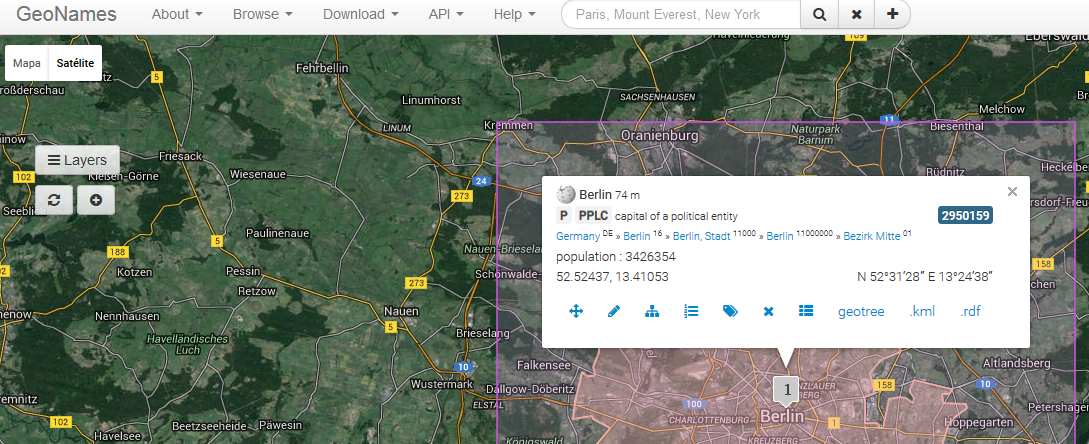
http://www.geonames.org/2950159
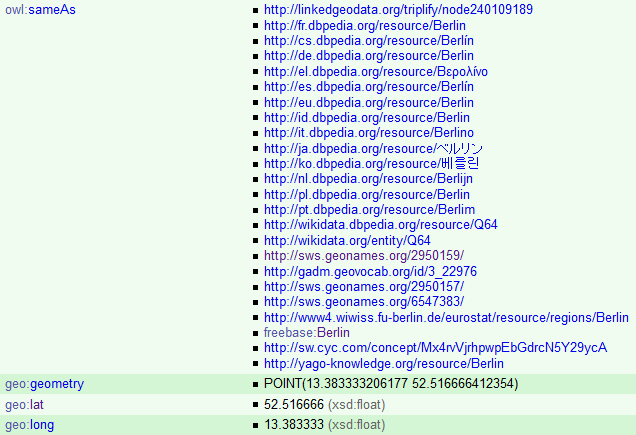
owl:sameAs
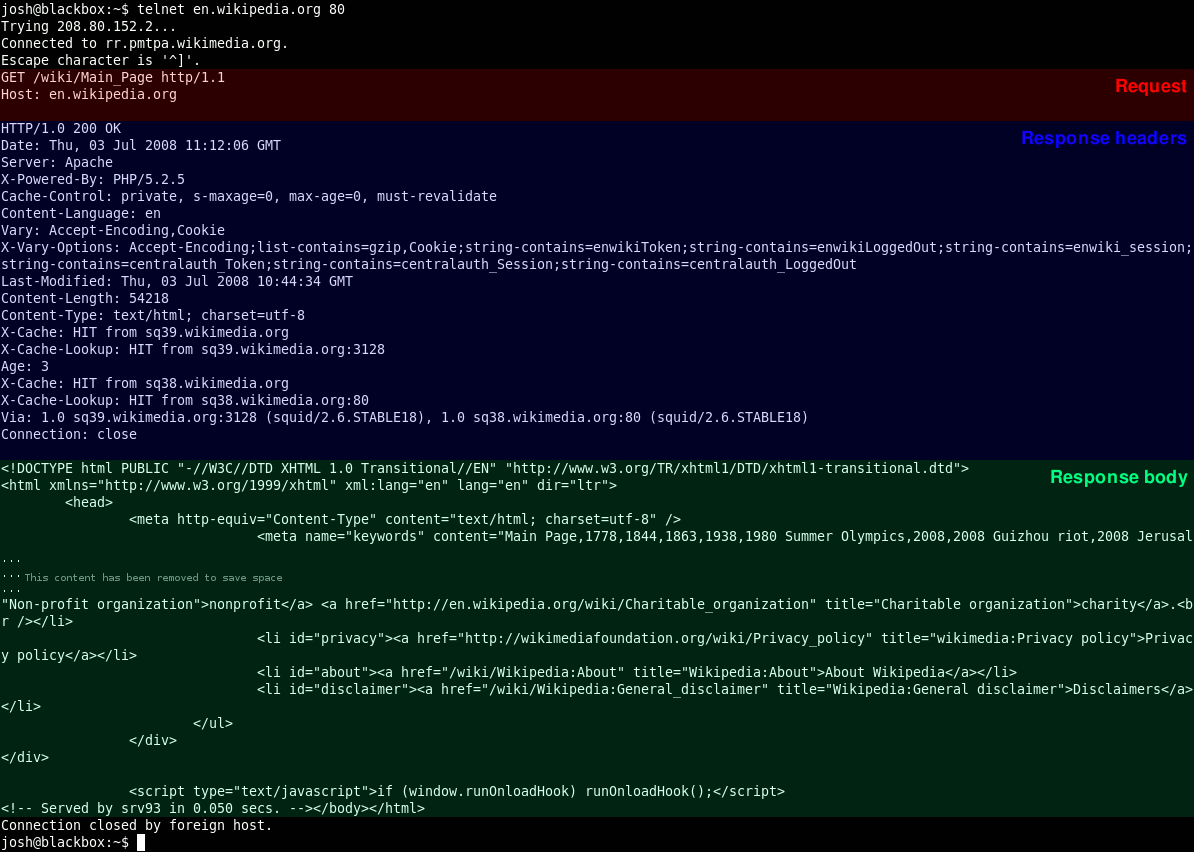
HTML
RDF
HTML
RDF
URIs = identificadores
Negociacion contenido (conneg)
curl -L -H "Accept: text/html" "http://dbpedia.org/resource/Berlin"

curl -L -H "Accept: application/rdf+xml" "http://dbpedia.org/resource/Berlin"

curl -L -H "Accept: text/html" "http://sws.geonames.org/2950159/"
curl -L -H "Accept: application/rdf+xml" "http://sws.geonames.org/2950159/"
URIs/URLs en Linked Data
URI identifica a entidad; URLs localizan diferentes representaciones (RDF, HTML, ...) de la entidad
Descripción de la entidad (RDF, HTML, ...) ≠ entidad
HTTP URI dereferenciable: cuando se busca una URI, deberia devolver una descripción adecuada del objeto que identifica esa URI
Negociación contenido

Negociación contenido

Negociación contenido

Linked Data
Publicar datos:
- Con "semántica" explícita
- Con enlaces
Semántica
RDF ofrece el triple, un modelo de datos explícito y homogéneo: una "frase" estándar que los ordenadores pueden "entender"

Enlaces
En el triple, cada entidad (sujeto, predicado, objeto) tiene una URI que lo identifica
Los datos son enlazados a otros datos a través de la Web, con enlaces explícitos

Grafo

Red global de datos enlazados

SPARQL
SELECT ?lugar ?nombre
WHERE {
?lugar <http://dbpedia.org/located_in> <http://gip.eus/donostia> .
?lugar rdfs:label ?nombre
}
SPARQL endpoint (Triple Store)
Consultas federadas
Integración
(SERVICE)
- URIs = "keys"
- Enlaces
OWL
Crear ontologías mediante Web Ontology Language
Ontología: "esquema" que describe el conocimiento sobre los datos
Tiene clases de individuos y define las condiciones para pertenecer a una clase
Es un lenguaje axiomatico con semantica precisa >> razonamiento automático
Algunas Triple Stores incluyen razonamiento automático en consultas
sujeto/objeto RDF >> rdf:type >> URI Clase OWL
Arquitectura Linked Data
Triple Store: almacena RDF
SPARQL endpoint: interfaz de consulta a Triple Store (humanos y máquinas)
Servidor Linked Data: sirve HTML o datos RDF mediante negociación de contenido
Triple store: Almacenar RDF, SPARQL endpoint
Servidor Linked Data: acceso web, negociación contenido

Arquitectura Linked Data

Interfaz datos
Interfaz para programadores/expertos en datos
Análisis complejos de los datos/nueva aplicaciones
SPARQL: Consultas complejas contra los datos, incluso combinando diferentes «bolas» de la nube Linked Open Data (datasets externos)
RDF: Crear programas autónomos que «naveguen» por los datos, recolectando datos (agentes)
Interfaz datos SPARQL
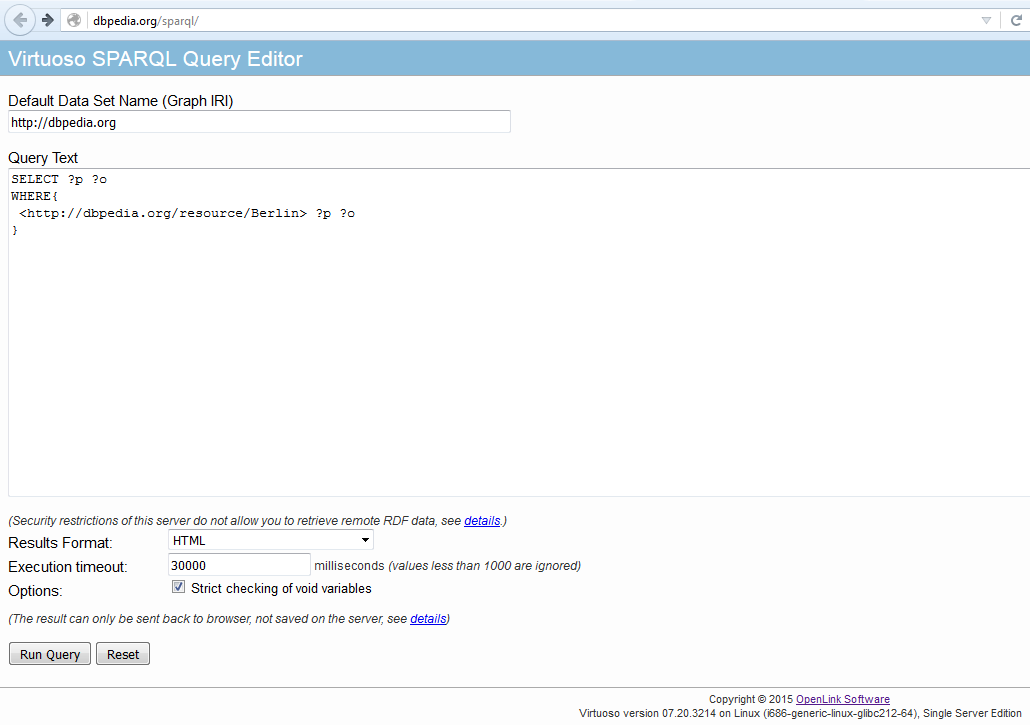
curl "http://dbpedia.org/sparql?query=SELECT+%3Fp+%3Fo%0D%0AWHERE+{%0D%0A<http%3A%2F%2Fdbpedia.org%2Fresource%2FBerlin>+%3Fp+%3Fo}"
curl "http://dbpedia.org/sparql?query=SELECT ?p ?o WHERE {<http://dbpedia.org/resource/Berlin> ?p ?o}"
Interfaz HTML
Interfaz para usuarios no expertos
Navegar por los datos publicados

Resultados
Integración en nube LOD
Enlaces a otros recursos Linked Data
La capacidad de los datos de «ser descubiertos» aumenta
Los datos son más interoperables a través de
a) Uso de triple RDF (modelo de datos común)
b) Ontologías comunes («esquema» común)
c) Uso de URIs para entidades (identificadores enlazables)
Beneficios para usuarios
Acceder a datos de manera más rica a través de la web (la web son los datos, no un documento que representa los datos)
Acceder a más datos, con enlaces más ricos («es parte de», «nació en», … ) a otros recursos: descubrimiento de nuevos datos
Encontrar datos de manera más precisa
Un ecosistema más rico de Apps, ya que es más fácil desarrollar Apps que integren datos
Beneficios para desarrolladores
Crear programas nuevos fácilmente: ej. visualizaciones especificas
Analizar los datos exhaustivamente, en relación a datos externos: ej. estadísticas locales vs estadísticas a nivel europeo
Integración de datos
Descubrimiento de nuevos datos
¿Por qué publicar datos en LD?
Enlaces al exterior:
Publicar solo nuestros datos, referencias al resto, no hay que replicar datos externos
Los datos externos se actualizan independientemente, y nuestro dataset va "a remolque" sin esfuerzo
¿Por qué publicar datos en LD?
Enlaces a nuestro dataset:
Es facil enlazar a nuestro dataset, ya que usamos HTTP URIs
Por lo tanto, aumenta la capacidad de nuestro dataset de ser descubierto mediante enlaces
¿Por qué publicar datos en LD?
Semántica:
El significado de nuestro datos es explícito y claro, debido a RDF (instancias) + OWL ("esquema")
Es "fácil" crear aplicaciones, incluyendo razonamiento automático (ej. agentes)
Open Authors
Proceso Open Authors



Publicar Linked Data
Publicar datos LD
1) Crear el dataset
Ontología OWL: reusar lo más posible de otras ontologías para interoperabilidad
Instancias RDF (triples)
2) Añadir enlaces a otros datasets
Manualmente o con herramientas como SILK, LIMES, Refine, ...
A nivel de instancias (owl:sameAs, predicados, ...) y a nivel de vocabulario (owl:equivalentClass, ...)
Publicar datos LD
3) Almacenar el dataset en triple store
4) Publicar el dataset mediante servidor web

Publicar datos LD

Publicar datos LD


Publicar datos LD
Linked Data

Ejercicio
Recrear todo el proceso de publicar nuestro dataset Linked Data en nuestro servidor, en una infraestructura ya preparada (Linked Data Server)
~# cd LSSW-UM-2016-2017/LinkedDataServer/blazegraph
~# java -server -Djetty.port=8081 -jar bigdata-bundled.jar
[Subir datos?]
~# cd LSSW-UM-2016-2017/LinkedDataServer/pubby
~# java -jar start.jar jetty.port=8080

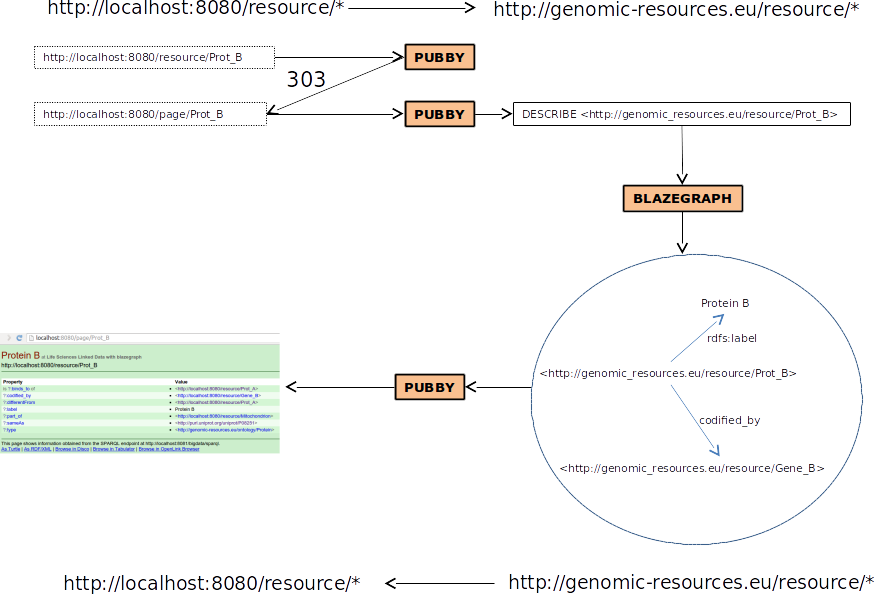
Linked Data
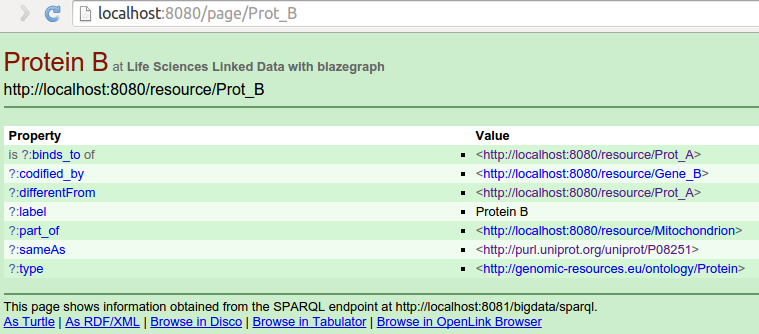
Linked Data
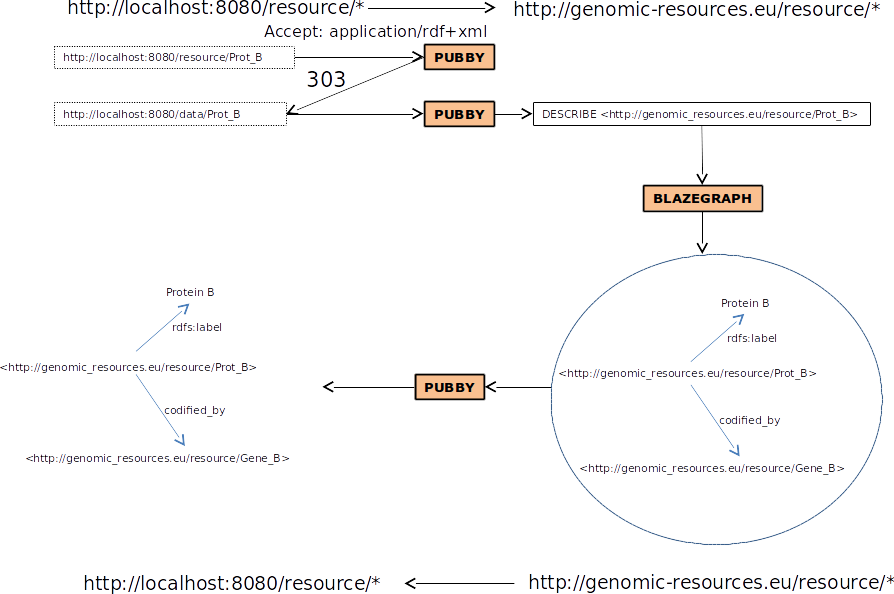
Negociación de contenido en terminal:
$ curl -L "http://localhost:8080/resource/Prot_B"
$ curl -L -H "Accept: application/rdf+xml" http://localhost:8080/resource/Prot_B
Configuracion Pubby
pubby/webapps/ROOT/WEB-INF/web.xml
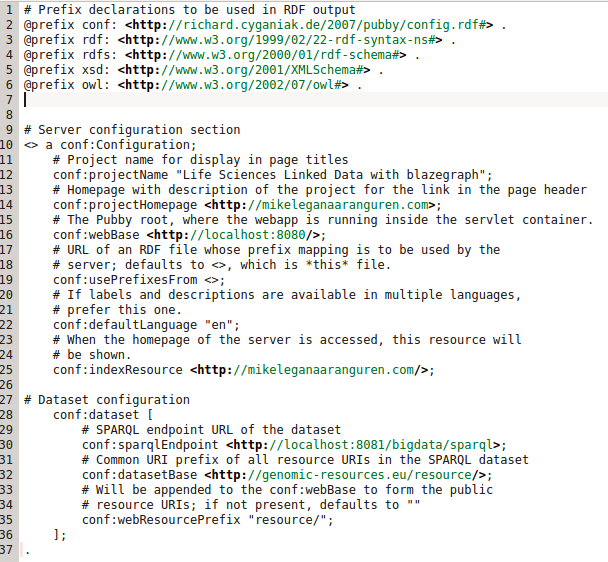
pubby/webapps/ROOT/WEB-INF/blazegraph-config.ttl
Crear un dataset nuevo en un archivo RDF (ttl?), con otras URIs (pocos triples)
Subirlo a blazegraph
Configurar pubby
Ejercicio
Servicios Web Semánticos
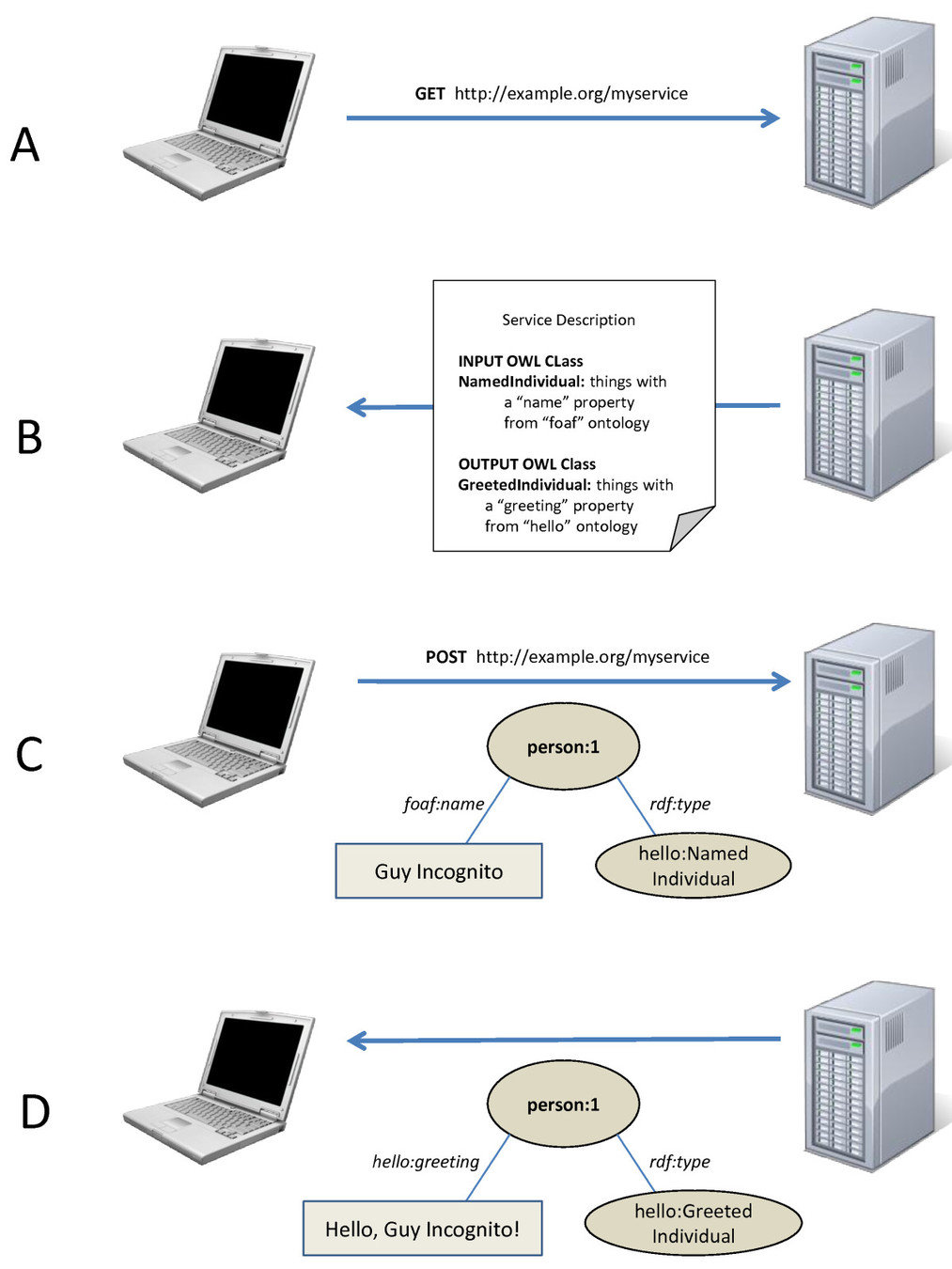

Given a set of UniProt proteins, which ones are related to PubMed abstracts containing the term "brain'', and what are they KEGG entries?
Q03164
Q9UKA4
Q8TDM6
Q9NQT8
Q12830
Q9HCM3
Q8TF72
Q5H8C1
Q9UGU0
B2RWN9
A4UGR9
B7ZML1
Given a set of UniProt proteins, which ones are related to PubMed abstracts containing the term "brain'', and what are they KEGG entries?

PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#>
PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#>
PREFIX sadi: <http://sadiframework.org/ontologies/predicates.owl#>
PREFIX lsrn: <http://purl.oclc.org/SADI/LSRN/>
SELECT ?protein ?label ?KEGG
WHERE {
?protein rdf:type lsrn:UniProt_Record .
?protein sadi:isEncodedBy ?KEGG .
?protein ?prot2hgnc ?hgnc .
?hgnc ?hgnc2omim ?omim .
?omim ?omim2pubmed ?pubmed .
?pubmed rdfs:label ?label .
FILTER (regex (?label, 'brain'))
}
curl -X POST --data-binary
@InputgetKEGGIDFromUniProt.rdf
http://sadiframework.org/services/getKEGGIDFromUniProt